BIOTECNOLOGÍA ESPAÑOLA / SPANISH BIOTECHNOLOGY
BIOLOGÍA SINTÉTICA: ASPECTOS CIENTÍFICOS Y SOCIALES
Carlos Alonso, Manuel Soto
Centro de Biología Molecular Severo Ochoa
Universidad Autónoma de Madrid
calonso@cbm.csic.es, msoto@cbm.csic.es
RESUMEN
El 20 de Mayo de 2010 Craig Venter anunció que se había creado el primer genoma sintético auto-replicativo capaz de controlar el comportamiento metabólico de las células huésped. La reacción al anuncio de Venter fue de sorpresa y asombro, pero levantó toda una gama de alarmas desde entusiasmos alumbrados a exclamaciones de incertidumbre: ¿Qué quería decir este anuncio? La biología entraba en la era digital y afirmaciones sobre potenciales riesgos y beneficios invadieron el mundo científico. ¿Estamos acercándonos a la “creación de vida” mediante diseño? ¿Se podría crear materia viva a partir de materia no viva? ¿Cuál es la diferencia entre materia viva y no-viva? ¿El organismo que se había generado por síntesis química era el que se había diseñado? El reto estaba en saber diseñar las condiciones para que los genomas y los huéspedes funcionaran al unísono. El reto es, por tanto, científico, pero lo es igualmente técnico. ¿Tenemos los conocimientos suficientes para diseñar las interacciones entre unidades biológicas capaces de generar organismos nuevos? ¿Podría la biología sintética jugar algún papel en la clínica?
SYNTHETIC BIOLOGY: SCIENTIFIC AND SOCIAL ASPECTS
ABSTRACT
On 20 May 2010, Craig Venter announced that he had created the first self-replicating synthetic genome capable of controlling the metabolic behaviour of host cells. The reaction to Venter’s announcement was surprise and amazement, but raised a range of alarms from enlightened enthusiasm to exclamations of uncertainty: What did this announcement mean? Biology had entered the digital age. Claims about potential risks and benefits invaded the scientific world. Were we approaching the “creation of life” by design? What is the difference between living and non-living matter? Was the organism generated by chemical synthesis the same as that which had been designed? The challenge was to find the conditions in which the genomes and the enucleated hosts could function in unison. Do we have enough knowledge to design interactions between biological units capable of generating new organisms? Scientists have used DNA to modify cells and organisms. In fact, a large industry has grown up based on recombinant DNA technology. Could synthetic biology play a clinical role?
Recibido: 09-06-2014; Aceptado: 14-07-2014.
Cómo citar este artículo/Citation: Alonso, C. y Soto, M. (2014). "Biología sintética: aspectos científicos y sociales". Arbor, 190 (768): a148. doi: http://dx.doi.org/10.3989/arbor.2014.768n4002
PALABRAS CLAVE: Vida; síntesis; complejidad; genomas; riesgos; DNA.
KEYWORDS: Life; synthesis; complexity; genomes; risks; DNA.
Copyright: © 2014 CSIC. Este es un artículo de acceso abierto distribuido bajo los términos de la licencia Creative Commons Attribution-Non Commercial (by-nc) Spain 3.0.
CONTENIDOS
INTRODUCCIÓN
Top
El 20 de Mayo de 2010 Craig Venter anunció que se había creado el primer genoma sintético auto-replicativo y que una vez insertado en el interior de una bacteria el genoma sintético era capaz de controlar el comportamiento metabólico del huésped. Años antes, en un día caluroso de verano y con todo el boato que se merecía, se puso de manifiesto que por primera vez se había logrado descifrar el código genético humano mediante la lectura de los 3 mil millones de letras que formaban la molécula de DNA. El anuncio de la lectura de la molécula de DNA humano ponía de manifiesto que se había cruzado un puente hacia una nueva era de la biología. Collins estableció que la nueva era de la biología no se restringía a un aumento en la cantidad de conocimientos sino que implicaba que se entraba en una etapa en la que mediante la lectura del DNA se podrían diseñar nuevos fármacos y se abría el paso a diseñar nuevos derroteros terapéuticos personalizados. El anuncio de Collins y de Venter fue noticia en todo el mundo e inundó los más diversos medios de comunicación.
En realidad la afirmación de que el siglo XXI sería el siglo de la biología no se deducía de una tesis más a o menos gratuita o iluminada sino que era consecuencia de la conjunción de un enorme cúmulo de conocimientos que hasta ese momento se habían ido esparciendo y cimentando durante la primera y segunda mitad del siglo 20. La verdadera transformación se centraba en el hecho de que a partir de este momento los conocimientos podían traducirse en instrumentos operativos. Ya en 1978 se había anunciado la llegada de una “nueva era de la biología” a la que se le dio el nombre de “sintética” porque se podrían sintetizar moléculas de DNA con objeto de diseñar genes. Restaba por saber si esos genes serían capaces de dirigir procesos metabólicos.
Para todos aquellos que han sido espectadores del maravilloso espectáculo que ofreció el desarrollo de la Biología en las décadas ´40-80’, el anuncio de Collins y Venter no era sino el fruto maduro de los conocimientos generados con anterioridad. Curiosamente, no se tenía conciencia de lo que podrían significar aunque se empezaba a vislumbrar que la vida era el resultado ordenado de la interacción de los elementos químicos. Se empezó a soñar que si se podía conocer el modo cómo los elementos químicos transferían la información e interaccionaban entre si, y que si se pudieran sintetizar químicamente tales elementos químicos, se podría diseñar la vida de una manera similar a como se diseña un edificio. Bastaba componer el plano y colocar las piezas según el diseño. Más aún, los comportamientos vitales se podrían controlar dado que los genes controlaban la fisiología del sistema mediante el control de las rutas metabólicas.
SIGNIFICADO DIGITAL DE LA BIOLOGÍA SINTÉTICA
Top
La reacción social al anuncio de Collins fue de sorpresa y asombro. La reacción al anuncio de Venter fue también de sorpresa y asombro, pero con otros tonos, y no se hizo esperar: se levantó toda una gama de alarmas desde entusiasmos alumbrados a exclamaciones de incertidumbre: ¿Qué quería decir este anuncio? ¿Significaría un cambio significativo en lo que entendíamos hasta este momento por Biología o simplemente era un anuncio más de los muchos a los que últimamente estamos acostumbrados a escuchar y que se olvidaría a los pocos días de su anuncio? Para algunos el anuncio de que la transferencia de unos genes a una bacteria podían alterar el fenotipo y hacer que el huésped desempeñase unas funciones determinadas, era la confirmación de que la vida no era sino el fruto del juego de relaciones digitales entre moléculas. También en biología entrábamos en la era digital como había anunciado la informática. Afirmaciones sobre potenciales riesgos y beneficios invadieron el mundo científico y no científico.
Aunque la barrera existente entre el mundo inorgánico y el orgánico, convertida en dogma durante muchos siglos, se había roto con la síntesis de la urea por Whöler, se pensaba que la barrera existente entre el mundo orgánico y el viviente parecía todavía subsistir y que subsistiría aun después de esta primera ruptura pues la materia viva y la materia no-viva pertenecían a ordenes ontológicos diferentes. No había posibilidad de paso de una esfera a otra. La ruptura de la barrera entre lo inorgánico y lo orgánico, efectuada por la síntesis de la urea, podría parecer un hecho insignificante cuando se le compara con la generación de una molécula de gran complejidad como el genoma sintetizado por Venter. Pero, en realidad no es así si nos trasladamos al momento en que Whöler, en el 1828, presentó sus experimentos. Otros investigadores habían fracasado en su intento y de hecho, en torno a la síntesis de la urea, se levantaron toda clase de especulaciones tanto científicas como filosóficas. ¿Estábamos ante el inicio científico de la alquimia?
¿Se podría crear materia viva a partir de materia no viva? ¿Cuál es la diferencia entre la materia viva y no-viva? ¿Los elementos que forman la materia viva serían diferentes a los elementos que formaban la materia no-viva? La lógica de la que emergían tales preguntas parecía aplastante puesto que ¿no era verdad que científicamente hablando había que suponer que el origen de la materia viva debería haber procedido de algo no vivo a menos que se admitiera que la materia viva había existido siempre junto a la no viva? Puesto que esta segunda afirmación no podía ser mantenida científicamente habría que suponer que se podría construir vida de no-vida. Pero ese no era el problema fundamental. Supongamos que se pudiera construir vida de no vida, la pregunta sería: Y ¿Cómo? Whöler dio el primer paso. De hecho, la ruptura de esta primera barrera ha dado lugar a una floreciente e industriosa revolución que ha invadido aún el mundo de la medicina para entrar en la era de la Farmacología y Biomedicina. Tanto influyó el anuncio de Venter que algunas mentes llegaron a ver en el tejido social vestigios de comportamientos de tipo biológico y que resortes de este tipo podrían influir en lo social. Así, el siglo 21 sería el siglo de la Biología e igualmente el siglo de la Antropología biológica al poder controlar con moléculas químicas el comportamiento humano. Lo que se había hecho con una bacteria, ¿podría hacerse con un huésped más complejo?
Aunque Venter utilizó la palabra “crear vida”, en el ambiente científico todos entendimos lo que en realidad había hecho o había querido decir: Venter había sintetizado un genoma capaz no solo de dirigir las funciones de la bacteria sino que podía auto-replicarse en su interior dado que el huésped con su maquinaria citoplasmática era capaz de expresar los genes presentes en el genoma sintetizado, y mediante su expresión era capaz de controlar su metabolismo. El genoma controlaba rutas metabólicas pero más aún, la bacteria se controlaba a sí misma utilizando ese genoma. Se podría, entonces, hablar, con toda propiedad, de la creación de un organismo nuevo no existente con anterioridad.
La pregunta en este caso sería: ¿El organismo que se había generado era el que se había diseñado? No puede olvidarse que el genoma se había insertado dentro de un sistema celular que, en términos de interacción macromolecular y de red de interacciones, es mucho más complejo que el dictado por el genoma nuevo generado por síntesis. El genoma, por si solo, no podría haber hecho “nada” sin la mediación del sustrato citoplasmático de la bacteria. Lo importante era que la información procedente del nuevo genoma sintético podía dirigir las rutas metabólicas de la célula huésped a la que se había insertado. De alguna manera se podía decir que la bacteria huésped adoptaba el nuevo genoma como suyo y el genoma adoptaba el citoplasma huésped como suyo. Las piezas biológicas para generar un organismo eran intercambiables. Así, la biología sintética se podría definir como el diseño y construcción de nuevos sistemas biológicos no naturales y el rediseño de sistemas biológicos naturales ya existentes para lograr desarrollar aplicaciones útiles. En este sentido la Biología sintética es una tecnología especializada en la construcción y unión de sistemas biológicos complejos (Endy, 2005Endy, D. (2005). "Foundations for engineering biology". Nature, 438, pp. 449-453, http://dx.doi.org/10.1038/nature04342.).
¿DÓNDE RADICA LA NOVEDAD DE LA BIOLOGÍA SINTÉTICA?
Top
Lo verdaderamente sorprendente era que mediante 1- la unión de bloques químicos elementales se había podido construir una molécula de DNA, que 2- al unirse los bloques la molécula transportaba información y que 3- al interaccionar con otras unidades de la bacteria las informaciones que trasportaba se convertían a su vez en unidades operativas. Hasta entonces los bloques de información venían dados. Lo más que se podía hacer con ellos era aislarlos del organismo donde estaban y ser modificados mediante las técnicas del DNA recombinante desarrollas décadas atrás (ver más adelante referente al desarrollo de vacunas).
El grave interrogante que se le planteaba al mundo científico radicaba en que mientras los códigos de información dados hasta entonces habían sido seleccionados a través de millones de años de evolución y se había testado la interacción entre las moléculas, seleccionando las moléculas informativas no tóxicas, los nuevos códigos generados mediante síntesis no había pasado por el cribado de la evolución y por consiguiente no se conocía si eran beneficiosos o no. Durante muchos años los científicos habían utilizado técnicas de DNA recombinante para diseñar piezas de información genética para insertarlas en otros genomas. Las piezas procedían de un organismo vivo. Los códigos que se habían seleccionado por evolución no deberían, en principio, ser deletéreos. ¿Podrían los genomas que se sintetizaran químicamente ejercer funciones hasta ahora no controladas e incontrolables cuando se enfrentaran con el medio ambiente que sí eran fruto de la evolución? La molécula de DNA que antes había sido sintetizada por un organismo vivo (la bacteria) ahora la sintetizaba una máquina.
El anuncio de Venter iba más allá de la simple síntesis química de un genoma y de la inserción del genoma en una bacteria. El anuncio puso de manifiesto que se podían conjuntar dos sistemas, uno de gran complejidad interactiva con otro de gran complejidad informativa. El problema era que si esta conjunción y diálogo no se llevaban a cabo con absoluta precisión se podría arruinar tanto el genoma sintetizado como el organismo en el que se insertó tal genoma. Habría que hacer la conjunción con absoluta precisión. No había datos que predijeran a priori el éxito de tales conjunciones. De hecho, no sabemos nada de los fracasos y posibles destrucciones de los genomas sintetizados y de las bacterias huésped. Sí sabemos que en un caso el resultado fue positivo y que si se puede dar un caso se pueden dar más y de hecho se ha confirmado que así es. El reto está en saber diseñar las condiciones para que los genomas y los huéspedes funcionen al unísono. El reto es, por tanto científico, pero lo es igualmente técnico. ¿Tenemos los conocimientos suficientes para diseñar las interacciones entre unidades biológicas capaces de generar organismos nuevos? Por el momento, se puede afirmar que todavía no.
Por tanto, dependiendo de lo que se entienda por vida no sería erróneo afirmar que mediante la conjunción de una información genética exógena y una red de rutas citoplasmáticas, ya existentes, se podría generar un sistema de interacciones entre moléculas que se auto-replicasen y que, por tanto, tuviesen las características fundamentales de lo que llamamos vida. En realidad si contemplamos la diversidad de formas de vida que existe en la naturaleza, tendremos que concluir que el concepto vida no es sino una rica metáfora utilizada para expresar la existencia de un fenómeno homeostático de gran complejidad. Si esto es así, la adjetivación del término vida estaría en función del genotipo y fenotipo del sustrato al que se le atribuye el término vida. No bastaría ni el genotipo ni el fenotipo. Diversidades de vida habían sido generadas por la Naturaleza millones de años antes. El ser humano empezaba a imitar la acción de la naturaleza porque empezaba a entender el modo cómo la Naturaleza lo había hecho. A modo de principiantes, los científicos habían utilizado el DNA para modificar células y organismos. De hecho ha crecido una gran industria basada en la tecnología del DNA recombinante. ¿Con la nueva tecnología basada en la biología sintética, –síntesis química de genomas y una apropiada conjunción de sistemas informativos-, crecería una industria al mismo nivel?
LA INGENIERÍA COMO ELEMENTO ESENCIAL DE LA BIOLOGÍA SINTÉTICA
Top
Teniendo en cuenta lo anteriormente expuesto se puede fácilmente entender que el fin de la Biología sintética es generar, mediante ingeniería de procesos, sistemas biológicos que cumplan funciones diseñadas de antemano. Para que tal fin se lleve a cabo es necesario cumplir dos condiciones: Ser capaz de construir un gen y aún un genoma de gran longitud a partir de sus componentes elementales y lograr que ejerza la función para la que había sido diseñado cuando se le coloca en un ambiente determinado. Mediante la síntesis química se ha logrado construir un gen de gran longitud y aún un genoma de una longitud mucho mayor. Pero, para que la Biología sintética fuera totalmente autónoma tendría que ser capaz de diseñar y construir el sistema complejo donde situar el genoma sintetizado. El primer paso está dado y conseguido. El segundo es mucho más difícil y por ahora está lejos de poder ser conseguido puesto que no conocemos las reglas del juego interactivo que se lleva a cabo entre miles de moléculas. Esto no limita en nada la trascendencia de la Biología Sintética sino que la coloca en el marco donde, por el momento, debe situarse.
El desarrollo, hace poco más de una década, de un sistema de dos redes de genes que funcionan como un interruptor y un oscilador puso en marcha la rápida aparición de la Biología sintética como una rama de la ingeniería biológica de utilidad práctica. Pronto se diseñaron y se construyeron circuitos genéticos cada vez más sofisticados inspirados en la complejidad de circuitos eléctricos (Danino et al., 2010Danino, T.; Mondragón Palomino, O.; Tsimring, L. y Hasty, J. (2010). "A synchronized quorum of genetic clocks". Nature, 463, pp. 326-330, http://dx.doi.org/10.1038/nature08753.; You, R. et al., 2004You, L.; Cox, R. S.; Weiss, R. y Arnold, F. H. (2004). "Programmed population control by cell-cell communication and regulated killing". Nature, 428, pp. 868-871, http://dx.doi.org/10.1038/nature02491.) o electrónicos. Lo mismo que las redes físicas, las redes génicas debían complementarse interaccionando al unísono. Los circuitos físicos generan energía. Los circuitos génicos son capaces de controlar el comportamiento celular y el fenotipo. Al fin y al cabo, los circuitos génicos también generan energía. A partir de estos desarrollos se han diseñado y construido circuitos cada vez más complejos y se han comenzado a utilizar estos sistemas en una variedad de entornos, incluyendo la clínica. Igualmente se ha tratado de explorar y explotar la modularidad genética mediante el diseño de rutas biológicas. Obviamente estamos en los albores del empleo de la Biología sintética dado que los circuitos que se han logrado diseñar capaces de desarrollar funciones específicas son todavía de muy limitada complejidad. En Biología los límites están definidos por la complejidad. Cuanto más complejos sean los circuitos interactivos mayor será la especificidad aunque también lo será la incertidumbre en cuanto a su funcionamiento y potencial riesgo. No se debe descartar la posibilidad de introducir en nano-máquinas moléculas de DNA capaces de adoptar conformaciones diferentes y servir como interruptores biológicos y aun de uso industrial. El final de la historia es diseñar funciones.
LA BIOLOGÍA SINTÉTICA EN LA CLÍNICA
Top
1. Resistencia a antibióticos
Dado, por ejemplo, que las bacterias se están volviendo resistentes a los antibióticos de forma cada vez más rápida es necesario diseñar estrategias que tengan por objeto mejorar el arsenal antimicrobiano así como mejorar la eficacia de los antibióticos existentes. Por ejemplo, se han diseñado virus específicos para atacar o debilitar cepas bacterianas resistentes a antibióticos mediante la interrupción de los mecanismos de defensa. Concretamente el fago T7, de naturaleza lítica, que expresa la dispersina B es capaz de eliminar el 99,9% de las células bacterianas resistentes a los antibióticos cuando son infectadas con el fago. También se han diseñado adyuvantes biológicos sintéticos basados en fagos (M13 no lítico) modificados con objeto de mejorar la eficacia de quinolonas, β-lactámicos y aminoglucósidos (Lu y Collins, 2009Lu, T.K. y Collins, J.J. (2009). "Engineered bacteriophage targeting gene networks as adjuvants for antibiotic therapy". Proceedings of the National Academy of Sciences of the United States of America, 106, pp. 4629-4634, http://dx.doi.org/10.1073/pnas.0800442106.) mediante la destrucción de las redes que regulan los mecanismos de defensa. El tratamiento con estos fagos da como resultado que estos antibióticos tengan una eficacia unas 5.000 veces mayor. También se ha descrito la construcción de genes sintéticos para la construcción de una red de genes que podría, en principio, provocar que una población de mosquitos modificados en el laboratorio pudiera difundir rápidamente la modificación genética a una población de campo y de esta forma reducir la capacidad de trasmisión de la infección.
2. Tratamiento de tumores
Otro campo donde la biología sintética puede tener un radio de acción importante es el tratamiento de tumores. A pesar del éxito de las terapias contra el cáncer, la radioterapia y la quimioterapia siguen produciendo un daño considerable en las células sanas de los individuos tratados. Por esa razón es perentorio diseñar sistemas que sean capaces de distinguir con precisión células sanas de enfermas. Para este fin mediante biología sintética se pueden diseñar bacterias capaces de reconocer, invadir y destruir células cancerosas (Anderson et al., 2006Anderson, C.; Clarke, E. J.; Arkin, A. P. y Voigt, C. A. (2006). "Environmentally controlled invasion of cancer cells by engineered bacteria". Journal of Molecular Biology, 355, pp. 619-627, http://dx.doi.org/10.1016/j.jmb.2005.10.076.). Como ejemplo, se puede citar la transferencia a Escherichiacoli de un gen capaz de sintetizar la proteína INV (invasina) que se une fuertemente a receptores de la integrina β1 de mamíferos. Otros sistemas de erradicación de células cancerosas diseñado mediante Biología sintética consiste en introducir en el huésped por vía intravenosa bacterias modificadas capaces de invadir células cancerosas que tuvieran como objeto identificar una ruta tumorogénica. Las bacterias se diseñaron para suprimir mediante iRNA la expresión de CTNNB1 (que codifica β1-catenina). Se eligió este sistema dado quela sobre-expresión de este gen se asocia al inicio de muchos tipos de cánceres de colon.
3. Modificación de la fauna microbiota y macrobiota
Otro de los campos donde la Biología sintética puede tener gran relevancia se refiere a la modificación de la fauna microbiota de organismos de alta complejidad. Esta fauna es un ecosistema compuesto de miles de microorganismos que regulan la fisiología de los organismos huésped. Curiosamente, el número de esos microorganismos supera el de las células del organismo residente por un factor de 10 a 100. Al ser bien tolerados estos microorganismos son potenciales vectores para implementar circuitos genéticos sintéticos y así corregir anomalías metabólicas. Por ejemplo, la bacteria Escherichia coli, que expresa altos niveles de CAI-1 y AI-2, se ha utilizado como vector para prevenir la infección del cólera mediante el diseño de una interacción específica entre los microbios del intestino (Duan y March, 2010Duan, F. y March, J.C. (2010). "Engineered bacterial communication prevents Vibrio cholerae virulence in an infant mouse model". Proceedings of the National Academy of Sciences of the United States of America, 107, pp. 11260-11264, http://dx.doi.org/10.1073/pnas.1001294107.). Durante la infección Vibrio cholerae secreta factores de virulencia CT a baja densidad. V. choleraedetecta los niveles de un auto-inductor 1 (CAI-1) y un auto-inductor 2 (IA-2). Cuando ambos inductores se expresan en un nivel alto cesa la expresión de los factores de virulencia. La ingesta de bacterias modificadas por ratones infectados redujo dramáticamente la expresión de los niveles de la toxina del cólera. Así, mediante la programación de circuitos génicos se pueden ejecutar procesos sofisticados que tras la implantación en un organismo pueden desempeñar funciones aplicables a una medicina personalizada. En el campo de la macrobiota se puede citar la generación de células troncales pluripotentes o células iPS a partir de células epiteliales mediante la construcción de redes génicas que les dotan de una capacidad de desarrollo y diferenciación específica y diseñada.
4. Generación de vacunas
A. Empleo del uso de vectores plasmídicos
El desarrollo de las técnicas biotecnológicas vivido en los últimos 20 años ha permitido realizar grandes avances en el diseño de nuevas vacunas. La primera gran aportación de la biotecnología ha sido el reemplazo de mezclas de antígenos por proteínas de los mismos organismos expresadas gracias a las técnicas del DNA recombinante. El caso más paradigmático es la vacuna contra la hepatitis B basada en las proteínas de la envuelta del virus (proteína HBVS, fundamentalmente) obtenidas de forma recombinante en levaduras o en cultivos de células de mamífero.
En la actualidad, la creación de vacunas basadas en proteínas recombinantes se encuentra en diferentes fases de investigación y desarrollo. Los sistemas más sencillos de producción de proteínas heterólogas son aquellos basados en el uso de bacterias tales como E.coli. Los vectores de expresión bacterianos son típicamente plásmidos que contienen uno o más orígenes de replicación y genes de selección. Las proteínas heterólogas se pueden obtener fusionadas a péptidos lo que permite una eficiente purificación por afinidad. Otra ventaja es la facilidad del crecimiento bacteriano en medios definidos de bajo coste y con posibilidad de ser escalados en procesos de producción. Como principal desventaja cabe destacar la incapacidad de las bacterias de generar muchas de las modificaciones necesarias para obtener las proteínas maduras tal y como se presentan al sistema inmunológico en los patógenos y que son necesarias para recrear la antigenicidad de las mismas. Otro problema adicional de los sistemas bacterianos es la posible contaminación de las proteínas heterólogas con endotoxinas, que por interaccionar con el receptor TLR-4 del sistema inmunológico innato puede generar respuestas indeseadas (Beutler y Rietschel, 2003Beutler, B. y Rietschel, E. T. (2003). "Innate immune sensing and its roots: the story of endotoxin". Nature Reviews. Immunology, 3, pp. 169-176, http://dx.doi.org/10.1038/nri1004.).
Como ejemplo de desarrollo de vacunas recombinantes frente a una infección causada por un virus se pueden citar los esfuerzos realizados en el campo de las vacunas contra Dengue. En este caso el candidato más destacado es el empleo de la glicoproteína E del virus. El objetivo de la inmunización con las vacunas basadas en la glicoproteína E es conseguir generar respuestas de anticuerpos neutralizantes, acompañadas además por la inducción de respuestas celulares mediadas por linfocitos T CD4+ y CD8+. Existen otros ejemplos de proteínas recombinantes expresadas en bacterias que, en la actualidad, se encuentran en proceso de investigación, de desarrollo o incluso comercializadas. Como muestra se pueden citar una vacuna basada en una proteína de Bucella abortus capaz de generar protección en modelos experimentales de ratón de esta patología, la proteína quimérica compuesta por tres antígenos de Leishmania (LEISH110) en fase 2 de su análisis como vacuna frente a leishmaniosis humana o la vacuna basada en la versión recombinante de la proteína A2 de Leishmania (Beaumier et al., 2013Beaumier, C. M.; Gillespie, P. M.; Hotez, P. J. y Bottazzi, M. E. (2013). "New vaccines for neglected parasitic diseases and dengue". Translational Research, 162, pp. 144-155, http://dx.doi.org/10.1016/j.trsl.2013.03.006.).
Las células eucariotas se han convertido en la alternativa razonable para la expresión de antígenos modificados. Por su versatilidad y otras ventajas pueden llegar a desplazar a los sistemas bacterianos en un futuro próximo. Dependiendo del sistema eucariota que se emplee los vectores presentarán diseños diferentes. Los más evolucionados están basados en levaduras. Habitualmente se emplean cepas de levaduras para adecuar los patrones de modificación de las proteínas a los realizados por las células de los hospedadores, como se ha descrito para las versiones recombinantes de la Neuraminidasa del virus influenza (Yang et al., 2012Yang, Y.L.; Chang, S.H.; Gong, X.; Wu, J. y Liu, B. (2012). "Expression, purification and characterization of low-glycosylation influenza neuraminidase in alpha-1,6-mannosyltransferase defective Pichiapastoris". Molecular Biology Reports, 39, pp. 857-864, http://dx.doi.org/10.1007/s11033-011-0809-z.). Estos vectores han permitido también obtener algunos de los antígenos en forma soluble que en sistemas procariotas se expresaban y aislaban en cuerpos de inclusión. Un ejemplo típico del uso de este tipo de plásmidos es el utilizado para generar vacunas experimentales frente a Leishmania empleando proteínas de la saliva de sus insectos vectores (Gomes et al., 2012Gomes, R.; Oliveira, F.; Teixeira, C.; Meneses, C.; Gilmore, D.C.; Elnaiem, D.E.; Kamhawi, S. y Valenzuela, J.G. (2012). "Immunity to sand fly salivary protein LJM11 modulates host response to vector-transmitted Leishmania conferring ulcer-free protection". Journal of Investigative Dermatology, 132, pp. 2735-2743, http://dx.doi.org/10.1038/jid.2012.205.).
Un interesante uso alternativo de estos plásmidos de expresión eucariota es su empleo como vacunas de DNA. Estas vacunas de tercera generación parten de las observaciones realizadas por Wolf y colaboradores (Wolff et al., 1990Wolff, J. A.; Malone, R.W.; Williams, P.; Chong, W.; Acsadi, G.; Jani, A. y Felgner, P.L. (1990). "Direct gene transfer into mouse muscle in vivo". Science, 247, pp. 1465-1468, http://dx.doi.org/10.1126/science.1690918.), donde describen que la inoculación intramuscular de plásmidos es capaz de transfectar miocitos y por lo tanto inducir una eficaz producción de proteínas heterólogas. Esta expresión puede generar tanto respuestas de anticuerpos como respuestas celulares, implicando tanto a los linfocitos CD4+ Th1 como CD8+. En la inducción de las respuestas observadas se han propuesto dos mecanismos que implican a células presentadoras de antígeno (Figura 1). La transfección directa de estas células provocan la producción intracelular de las proteínas heterólogas que son procesadas para su presentación en el complejo mayor de histocompatibilidad de tipo I (generando respuestas citotóxicas, CD8+ y de tipo II, estimulando la diferenciación y expansión de células CD4+. La interacción de las moléculas del plásmido con el receptor TLR-9 induce la liberación de IL-12, que promueve la diferenciación de los linfocitos T CD4+ hacia el fenotipo Th1. La complejidad de las respuestas generadas las convierte en candidatos para el tratamiento de un gran número de enfermedades causadas por agentes infecciosos. Los vectores diseñados para estas vacunas no poseen genes de selección y tienen optimizado el uso de codones en las regiones codificantes de las proteínas vacunales. La administración de adyuvantes junto a las proteínas vacunales, las estrategias de “prime-boost” y la secreción de proteínas heterólogas o su fusión con secuencias, que las dirigen hacia células presentadoras de antígenos, intentan maximizar los efectos protectores de las vacunas (Gurunathan et al., 2000Gurunathan, S.; Klenman, D. M. y Seder, R. A. (2000). "DNA vaccines: immunology, application, and optimization". Annual Review of Immunology, 18, pp. 927-974, http://dx.doi.org/10.1146/annurev.immunol.18.1.927.; Cayabyab et al., 2012Cayabyab, M. J.; Macovei, L. y Campos-Neto, A. (2012). "Current and novel approaches to vaccine development against tuberculosis". Frontiers in Cellular and Infection Microbiology, 2, p. 154. http://dx.doi.org/10.3389/fcimb.2012.00154.).
|
Figura 1. Proceso de transfección de miocitos o de células presentadoras de antígeno (APC) con ADN
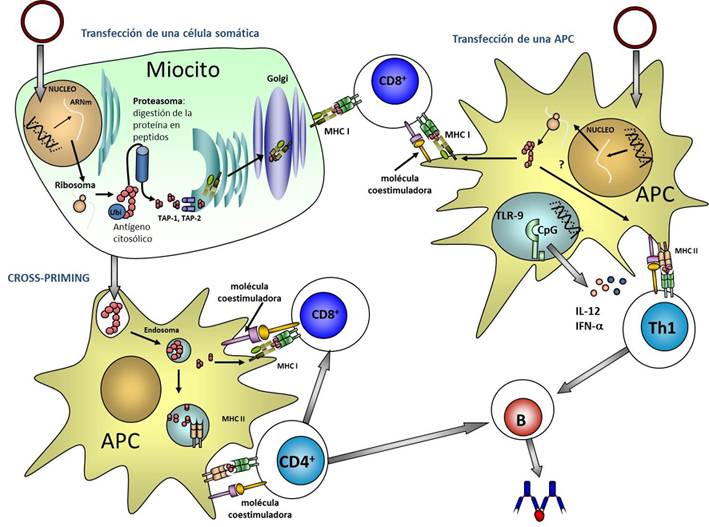
Abreviaturas: MHCI, Complejo Mayor de Histocompatilidad Clase I; MHCII, Complejo Mayor de Histocompatilidad Clase II; CD4+, linfocito T que expresa la proteína CD4; CD8+, linfocito T que expresa la proteína CD8; B, linfocito tipo B; Th1, linfocito tipo Th1; IL-12, interleukina 12; IFNα, interferón tipo alfa; TAP1 y TAP2, transportador asociado con el procesamiento de antígenos; TRL-9, receptor tipo Toll; CpG,dinucleótidoCpG; ARNm, RNA mensajero.
[Descargar tamaño completo]
| |
B. Un ejemplo del desarrollo en España de vacunas contra Leishmania
Tras la infección por parásitos y muy particularmente tras la infección por Leishmania bien en perros o humanos determinadas proteínas ribosomales son partículas inmuno-dominantes. En concreto las proteínas ribosomales P0, P2a y P2b son ejemplo de antígenos generados mediante infección por parásitos intracelulares. De hecho después de una infección visceral tanto en perros como humanos se generan fuertes respuestas inmunes contra estas proteínas (Coelho et al., 2009Coelho, E. A.; Ramírez, L.; Costa, M.A.; Coelho, V.T.; Martins, V.T.; Chávez-Fumagalli, M.A.; Oliveira, D.M.; Tavares, C.A.; Bonay, P.; Gómez Nieto, C.; Abánades, D.R.; Alonso, C. y Soto, M. (2009). "Specific serodiagnosis of canine visceral leishmaniasis using Leishmania species ribosomal protein extracts". Clinical and Vaccine Immunology, 16, pp. 1774-1780, http://dx.doi.org/10.1128/CVI.00295-09.; Ramírez et al., 2013Ramírez, L.; Santos, D.M.; Souza, A.P.; Coelho, E.A.; Barral, A.; Alonso, C.; Escutia, M.R.; Bonay, P.; Oliveira, C.I. y Soto, M. (2013). "Evaluation of immune responses and analysis of the effect of vaccination of the Leishmania major recombinant ribosomal proteins L3 or L5 in two different murine models of cutaneous leishmaniasis". Vaccine, 31, pp. 1312-1319, http://dx.doi.org/10.1016/j.vaccine.2012.12.071.; Soto et al., 1995Soto, M.; Requena, J.M.; Quijada, L.; Ángel, S.O.; Gómez, L.C.; Guzmán, F.; Patarroyo, M.E. y Alonso, C (1995). "During active viscerocutaneous leishmaniasis the anti-P2 humoral response is specifically triggered by the parasite P proteins". Clinical and Experimental Immunology, 100, pp. 246-252, http://dx.doi.org/10.1111/j.1365-2249.1995.tb03661.x.). Curiosamente, a pesar de que estas proteínas están muy conservadas a lo largo de la evolución tanto en parásitos como perros o humanos los anticuerpos se generan contra secuencias específicas de las proteínas del parásito sin haberse observado reactividad cruzada. Algunas de estas proteínas son antigénicas en pacientes con enfermedades autoinmunes. Los epítopos B se sitúan en las partes más variables de P2a, P2b y P0 lo que explica que la respuesta inmune vaya dirigida contra esas zonas. La proteína P0 se ha empleado para el desarrollo de vacunas contra la forma cutánea tanto en ratones susceptibles BALB/c como resistentes (C57BL/6). Es interesante observar que en BALB/C la inmunización con un vacuna de DNA basada en P0 o con la proteína P0 combinada con un adyuvante induce protección parcial contra la infección por L. major. Una vacuna de este tipo induce protección total contra L. major en ratones 57BL/6. Se ha observado igualmente que la vacunación de ratones con una mezcla de proteínas ribosomales induce respuestas de tipo Th1 junto a la generación de respuestas protectivas y se correlaciona con protección contra L. major (Iborra et al., 2008Iborra, S.; Parody, N.; Abánades, D.R.; Bonay, P.; Prates, D.; Novais, F. O.; Barral-Neto, M.; Alonso, C. y Soto, M. (2008). "Vaccination with the Leishmania major ribosomal proteins plus CpG oligodeoxynucleotides induces protection against experimental cutaneous leishmaniasis in mice". Microbes and Infection, 10, pp. 1133-1141, http://dx.doi.org/10.1016/j.micinf.2008.06.002.), L. amazonensis o L. Chagasi (Chávez-Fumagalli et al., 2010Chávez-Fumagalli, M.A.; Costa, M.A.; Oliveira, D.M.; Ramírez, L.; Costa, L.E.; Duarte, M.C.; Martins, V.T.; Oliveira, J.S.; Olortegui, C.C.; Bonay, P.; Alonso, C.; Tavares, C.A.; Soto, M. y Coelho, E.A. (2010). "Vaccination with the Leishmania infantum ribosomal proteins induces protection in BALB/c mice against Leishmania chagasi and Leishmania amazonensis challenge". Microbes and Infection, 12, pp. 967-977, http://dx.doi.org/10.1016/j.micinf.2010.06.008.). La protección observada en el modelo BALB/c-L. major fue tan robusta que resistió una segunda infección (Ramirez et al., 2013). Además, la proteína S4 de L. braziliensis es reconocida por un clon T derivado de un paciente humano resistente a la infección DTH positivo. Esto indica que el reconocimiento por el sistema inmunológico de algunas proteínas ribosomales no está necesariamente relacionado con la progresión de la enfermedad. También se ha descrito que dos proteínas ribosomales LmL3 y LmL5 generan respuestas protectoras contra la infección por L. major y L. braziliensis en BALB/c. Una proteína quimérica basada en determinantes antigénicos H2A, P2a, P2b y P0 protege contra infección por Leishmania visceral en perros (Molano et al., 2003Molano, I.; Alonso, M. G.; Mirón, C.; Redondo, E.; Requena, J. M.; Soto, M.; Nieto, C. G. y Alonso, C. (2003). "Leishmania infantum multi-component antigenic protein mixed with live BCG confers protection to dogs experimentally infected with L. infantum". Veterinary Immunology and Immunopathology, 92, pp. 1-13, http://dx.doi.org/10.1016/S0165-2427(02)00315-X.; Carcelen et al., 2009Carcelén, J.; Iniesta, V.; Fernández Cotrina, J.; Serrano, F.; Parejo, J. C.; Corraliza, I.; Gallardo Soler, A.; Marañón, F.; Soto, M.; Alonso, C. y Gómez Nieto, C. (2009). "The chimerical multi-component Q protein from Leishmania in the absence of adjuvant protects dogs against an experimental Leishmania infantum infection". Vaccine, 27, pp. 5964-5973, http://dx.doi.org/10.1016/j.vaccine.2009.07.069.).
C. Generación de antígenos de diseño mediante la síntesis de genes sintéticos
En el gran cambio que se está llevando a cabo, en el entorno del desarrollo de vacunas, la Biología sintética puede desempeñar un papel crucial. Como se puede intuir ello es debido a poder sintetizar genes quiméricos de gran longitud y poder expresarlos en sistemas bacterianos, levaduras, células de insectos o sistemas vivos bien mediante administración directa del gen en forma de DNA (ver apartados anteriores) o mediante la administración de las proteínas expresadas. Así, se puede construir un gen quimérico en cuanto que contiene fragmentos de varias proteínas existentes o aún no existentes en la naturaleza. Al no existir en la naturaleza tal gen quimérico habría sido imposible su obtención. En la actualidad el diseño quimérico se hace tan fácilmente como escribir una frase en una hoja de papel. Basta saber colocar de forma apropiada los pares de bases uno detrás de otro en un orden tal que al ser leída por la maquinaria biológica genere una información precisa. Por eso lo verdaderamente importante es no solo concatenar las bases sino construir una frase que tenga significado, es decir un gen funcional en un contexto biológico determinado. Esta idea ya existía con anterioridad a la era de la Biología sintética, propiamente dicha, pero no había método para poderla llevar a la práctica. La gran revolución es que la construcción se puede hacer y además de forma sencilla. Ya existen máquinas que pueden generar esas concatenaciones. El arte está en el diseño y el reto está en saber cómo colocar las letras (elementos químicos) para que una vez insertado en un sistema vivo pueda generar una proteína funcional, antigénica e inmunogénica. Pero el reto es todavía mayor: No basta con construir el gen sino en saber cómo aislar la proteína codificada de forma que sea soluble, que sea estable dado que lo más probable es que la proteína diseñada no sea ni estable ni soluble, y que sea operativa. Hay que remontar un problema para el que por el momento no existe una solución general. Estabilidad y solubilidad son dos requisitos que se pueden considerar sine que non para el éxito de una vacuna. Por el momento las soluciones para lograr estabilidad y solubilidad son ad casum. Por eso la biología sintética sin una ingeniería biológica adecuada sería de utilidad limitada. Existen algoritmos para examinar (a) los grados de solubilidad y estabilidad, (b) el carácter hidrofóbico e hidrofílico, (c) la identificación de sitios de ruptura dentro de la proteína y (d) el diseño de epítopos de clase HLA I y II. Pero hay que señalar que todos estos algoritmos solo son indicativos. La prueba de fuego está en la demostración de que lo diseñado ejerce la función biológica predeterminada. En principio, no se puede excluir a priori que la proteína sintetizada quimérica no tenga alguna propiedad tóxica ya que con anterioridad nunca se había relacionado con un elemento vivo ni había pasado el tamiz de la evolución.
5. Aspectos sociales y éticos de la Biología sintética
Teniendo en cuenta que se trata de un campo de desarrollo nuevo con enormes potencialidades es necesario asegurar de antemano que a medida que se desarrolla el potencial de la Biología sintética se controle la razón beneficio/riesgo teniendo en cuenta que sin riesgos no hay beneficios que merezcan la pena. Hasta el momento los potenciales riesgos de la Biología sintética son solo prospectivos. Es probable que el principal riesgo que necesita ser supervisado se centre en la introducción de organismos nuevos en el medio ambiente, el análisis de cómo van a interactuar con él y la generación de genes sintéticos con efectos deletéreos tanto a nivel de DNA como de la proteína que codifican. Muchas de las directrices existentes para los organismos modificados genéticamente se podrían aplicar a la Biología sintética. Sin embargo pueden existir riesgos específicos que surjan no del producto final sintetizado sino de sus procesos de investigación y desarrollo. En este como en tantos otros campos se recomienda una actitud proactiva al mismo tiempo que recelosa. No existe el riesgo cero, ni son previsibles todos los potenciales riesgos pero precisamente por eso debe de forma periódica efectuarse una evaluación de los riesgos.
Desde un punto de vista ético, y dada la inmediata globalización de los conocimientos científicos, uno de los primeros pasos que es necesario dar para que la biología sintética tenga éxito desde un punto de vista social es diseñar sistemas de evaluación del riesgo derivado de la intercomunicación y asegurar el control de la exactitud de los datos diseminados. Los conocimientos científicos pueden ayudar a la humanidad a resolver graves problemas, sobre todo los referidos a la salud, pero invariablemente también pueden introducir riesgos y a menudo a generar preocupaciones y temores. Junto a estos beneficios está la obligación de considerar con la mayor precisión posible los potenciales peligros. Por eso el principio de precaución aplicado al uso de la Biología sintética ha ido adquiriendo fuerza cada vez mas destacada. No olvidemos que al hacer Biología Sintética nos movemos en un entorno de gran incertidumbre y que los objetivos son en realidad muy ambiciosos: generar una serie de instrumentos biológicos estándar de tipo universal que tengan características específicas de manera que puedan combinarse formando unidades que pueden no existir en la naturaleza. El ideal sería poder trasformar los procesos biológicos en procesos digitales más sencillos que pudieran ser adaptados a la industria de forma rutinaria y mecánica.
Desde un punto de vista sociológico cinco son los principios más relevantes que se deben tener en cuenta: 1- Beneficencia pública, que se centra en la obligación de promover actividades individuales y colectivas, incluyendo la investigación científica y biomédica. Estas actividades deben tener como objeto promover el bienestar público estando atentos a los riesgos. Por tanto el ideal de la beneficencia pública reside en implementar políticas para maximizar los beneficios públicos y reducir al mínimo los daños a la comunidad. 2- Gestión responsable, que se centra en pensar y actuar colectivamente en beneficio de la comunidad. Es importante destacar que se ha de tener una prudente vigilancia y que se han de establecer procesos de evaluación antes y después de que los proyectos se lleven a cabo. Una gestión responsable incluirá implementar mecanismos para limitar el uso de determinadas tecnologías cuando sea necesario. 3- Garantizar una autonomía y Libertad intelectual junto a responsabilidad. La ciencia, como conocimiento, desarrollo y tecnología, está en función de la libertad intelectual junto a la responsabilidad. Si bien muchas de las tecnologías emergentes suscitan preocupaciones por el hecho de que se pueden usar tanto para bien como para mal, es cierto que los temores existentes no justifican en muchos casos la limitación de la libertad intelectual. 4- Garantizar una deliberación pública, el principio de la deliberación pública va dirigida a la toma de decisiones que tienen en cuenta puntos de vista opuestos junto a la activa participación de los individuos involucrados. 5- El principio de justicia, con objeto de dirigir e identificar políticas públicas que aseguren que las nuevas tecnologías se desarrollen de forma responsable de manera que los beneficios se distribuyan de forma equitativa. En este sentido no se puede dejar de lado la cooperación internacional y el diálogo dado que las barreras biológicas se han estrechado cada vez más.
No cabe duda que del mundo inorgánico se ha pasado al orgánico, del mundo orgánico se ha pasado al molecular biológico y del mundo molecular biológico vivo se ha pasado al mundo molecular biológico sintético que tiene las características de lo vivo. A veces las utopías y las distopías parecen ser el único escenario. Hay que descubrir qué hay de realidad en las utopías y las distopías. Termino con las palabras de W. Wagner, Commission Vice Chair and President of Emory University: “We strongly support an open dialogue between DIY (Do it Yourself) groups and the government as we go forward so that scientists and government can discuss the research constraints necessary to protect public safety as the field continues to evolve.”
AGRADECIMIENTOS
Top
Ministerio de Ciencia e Innovación FIS PI11/00095; Instituto de Salud Carlos III “Network of Tropical Diseases Research” (VI P I+D+I 2008-2011, ISCIII-Subdirección General de Redes y Centros de Investigación Cooperativa (RD12/0018/0009)). Ayuda institucional de la Fundación Ramón Areces al CBMSO.
BIBLIOGRAFÍATop
|
| ○ | Anderson, C.; Clarke, E. J.; Arkin, A. P. y Voigt, C. A. (2006). "Environmentally controlled invasion of cancer cells by engineered bacteria". Journal of Molecular Biology, 355, pp. 619-627, http://dx.doi.org/10.1016/j.jmb.2005.10.076 |
| ○ | Beaumier, C. M.; Gillespie, P. M.; Hotez, P. J. y Bottazzi, M. E. (2013). "New vaccines for neglected parasitic diseases and dengue". Translational Research, 162, pp. 144-155, http://dx.doi.org/10.1016/j.trsl.2013.03.006 |
| ○ | Beutler, B. y Rietschel, E. T. (2003). "Innate immune sensing and its roots: the story of endotoxin". Nature Reviews. Immunology, 3, pp. 169-176, http://dx.doi.org/10.1038/nri1004 |
| ○ | Carcelén, J.; Iniesta, V.; Fernández Cotrina, J.; Serrano, F.; Parejo, J. C.; Corraliza, I.; Gallardo Soler, A.; Marañón, F.; Soto, M.; Alonso, C. y Gómez Nieto, C. (2009). "The chimerical multi-component Q protein from Leishmania in the absence of adjuvant protects dogs against an experimental Leishmania infantum infection". Vaccine, 27, pp. 5964-5973, http://dx.doi.org/10.1016/j.vaccine.2009.07.069 |
| ○ | Cayabyab, M. J.; Macovei, L. y Campos-Neto, A. (2012). "Current and novel approaches to vaccine development against tuberculosis". Frontiers in Cellular and Infection Microbiology, 2, p. 154. http://dx.doi.org/10.3389/fcimb.2012.00154 |
| ○ | Chávez-Fumagalli, M.A.; Costa, M.A.; Oliveira, D.M.; Ramírez, L.; Costa, L.E.; Duarte, M.C.; Martins, V.T.; Oliveira, J.S.; Olortegui, C.C.; Bonay, P.; Alonso, C.; Tavares, C.A.; Soto, M. y Coelho, E.A. (2010). "Vaccination with the Leishmania infantum ribosomal proteins induces protection in BALB/c mice against Leishmania chagasi and Leishmania amazonensis challenge". Microbes and Infection, 12, pp. 967-977, http://dx.doi.org/10.1016/j.micinf.2010.06.008 |
| ○ | Coelho, E. A.; Ramírez, L.; Costa, M.A.; Coelho, V.T.; Martins, V.T.; Chávez-Fumagalli, M.A.; Oliveira, D.M.; Tavares, C.A.; Bonay, P.; Gómez Nieto, C.; Abánades, D.R.; Alonso, C. y Soto, M. (2009). "Specific serodiagnosis of canine visceral leishmaniasis using Leishmania species ribosomal protein extracts". Clinical and Vaccine Immunology, 16, pp. 1774-1780, http://dx.doi.org/10.1128/CVI.00295-09 |
| ○ | Danino, T.; Mondragón Palomino, O.; Tsimring, L. y Hasty, J. (2010). "A synchronized quorum of genetic clocks". Nature, 463, pp. 326-330, http://dx.doi.org/10.1038/nature08753 |
| ○ | Duan, F. y March, J.C. (2010). "Engineered bacterial communication prevents Vibrio cholerae virulence in an infant mouse model". Proceedings of the National Academy of Sciences of the United States of America, 107, pp. 11260-11264, http://dx.doi.org/10.1073/pnas.1001294107 |
| ○ | Endy, D. (2005). "Foundations for engineering biology". Nature, 438, pp. 449-453, http://dx.doi.org/10.1038/nature04342 |
| ○ | Gomes, R.; Oliveira, F.; Teixeira, C.; Meneses, C.; Gilmore, D.C.; Elnaiem, D.E.; Kamhawi, S. y Valenzuela, J.G. (2012). "Immunity to sand fly salivary protein LJM11 modulates host response to vector-transmitted Leishmania conferring ulcer-free protection". Journal of Investigative Dermatology, 132, pp. 2735-2743, http://dx.doi.org/10.1038/jid.2012.205 |
| ○ | Gurunathan, S.; Klenman, D. M. y Seder, R. A. (2000). "DNA vaccines: immunology, application, and optimization". Annual Review of Immunology, 18, pp. 927-974, http://dx.doi.org/10.1146/annurev.immunol.18.1.927 |
| ○ | Iborra, S.; Parody, N.; Abánades, D.R.; Bonay, P.; Prates, D.; Novais, F. O.; Barral-Neto, M.; Alonso, C. y Soto, M. (2008). "Vaccination with the Leishmania major ribosomal proteins plus CpG oligodeoxynucleotides induces protection against experimental cutaneous leishmaniasis in mice". Microbes and Infection, 10, pp. 1133-1141, http://dx.doi.org/10.1016/j.micinf.2008.06.002 |
| ○ | Lu, T.K. y Collins, J.J. (2009). "Engineered bacteriophage targeting gene networks as adjuvants for antibiotic therapy". Proceedings of the National Academy of Sciences of the United States of America, 106, pp. 4629-4634, http://dx.doi.org/10.1073/pnas.0800442106 |
| ○ | Molano, I.; Alonso, M. G.; Mirón, C.; Redondo, E.; Requena, J. M.; Soto, M.; Nieto, C. G. y Alonso, C. (2003). "Leishmania infantum multi-component antigenic protein mixed with live BCG confers protection to dogs experimentally infected with L. infantum". Veterinary Immunology and Immunopathology, 92, pp. 1-13, http://dx.doi.org/10.1016/S0165-2427(02)00315-X |
| ○ | Ramírez, L.; Santos, D.M.; Souza, A.P.; Coelho, E.A.; Barral, A.; Alonso, C.; Escutia, M.R.; Bonay, P.; Oliveira, C.I. y Soto, M. (2013). "Evaluation of immune responses and analysis of the effect of vaccination of the Leishmania major recombinant ribosomal proteins L3 or L5 in two different murine models of cutaneous leishmaniasis". Vaccine, 31, pp. 1312-1319, http://dx.doi.org/10.1016/j.vaccine.2012.12.071 |
| ○ | Soto, M.; Requena, J.M.; Quijada, L.; Ángel, S.O.; Gómez, L.C.; Guzmán, F.; Patarroyo, M.E. y Alonso, C. (1995). "During active viscerocutaneous leishmaniasis the anti-P2 humoral response is specifically triggered by the parasite P proteins". Clinical and Experimental Immunology, 100, pp. 246-252, http://dx.doi.org/10.1111/j.1365-2249.1995.tb03661.x |
| ○ | Wolff, J. A.; Malone, R.W.; Williams, P.; Chong, W.; Acsadi, G.; Jani, A. y Felgner, P.L. (1990). "Direct gene transfer into mouse muscle in vivo". Science, 247, pp. 1465-1468, http://dx.doi.org/10.1126/science.1690918 |
| ○ | Yang, Y.L.; Chang, S.H.; Gong, X.; Wu, J. y Liu, B. (2012). "Expression, purification and characterization of low-glycosylation influenza neuraminidase in alpha-1,6-mannosyltransferase defective Pichiapastoris". Molecular Biology Reports, 39, pp. 857-864, http://dx.doi.org/10.1007/s11033-011-0809-z |
| ○ | You, L.; Cox, R. S.; Weiss, R. y Arnold, F. H. (2004). "Programmed population control by cell-cell communication and regulated killing". Nature, 428, pp. 868-871, http://dx.doi.org/10.1038/nature02491 |
|