SEGURIDAD ALIMENTARIA / FOOD SAFETY
AMINAS BIÓGENAS EN ALIMENTOS: MÉTODOS MOLECULARES PARA LA DETECCIÓN E IDENTIFICACIÓN DE BACTERIAS PRODUCTORAS
Beatriz del Río
Instituto de productos lácteos de Asturias. Consejo Superior de Investigaciones Científicas.
beadelrio@ipla.csic.es
ORCID iD: https://orcid.org/0000-0001-8107-1975
Begoña Redruello
Instituto de productos lácteos de Asturias. Consejo Superior de Investigaciones Científicas.
bredruel@ipla.csic.es
ORCID iD: https://orcid.org/0000-0003-1787-5594
María Fernández
Instituto de productos lácteos de Asturias. Consejo Superior de Investigaciones Científicas.
mfernandez@ipla.csic.es
ORCID iD: https://orcid.org/0000-0002-4413-6861
Víctor Ladero
Instituto de productos lácteos de Asturias. Consejo Superior de Investigaciones Científicas.
ladero@ipla.csic.es
ORCID iD: https://orcid.org/0000-0002-7613-3745
Miguel A. Álvarez
Instituto de productos lácteos de Asturias. Consejo Superior de Investigaciones Científicas.
maag@ipla.csic.es
ORCID iD: https://orcid.org/0000-0001-9607-7480
RESUMEN
Las aminas biógenas son compuestos nitrogenados de pequeño tamaño con actividad biológica que se forman por la descarboxilación enzimática de ciertos aminoácidos. Las aminas biógenas se encuentran presentes en todos los seres vivos, en los que participan en procesos biológicas de gran importancia. Sin embargo, debido al metabolismo de algunos microorganismos, estos compuestos se pueden acumular en alimentos en concentraciones elevadas, constituyendo un riesgo para la salud de los consumidores. Para que las aminas biógenas alcancen estas concentraciones elevadas en los alimentos se requiere, como condición indispensable, la presencia de microrganismos productores, por lo que se han desarrollado diferentes métodos para detectar la presencia de los mismos. Entre estos métodos, aquellos basados en técnicas independientes de cultivo, como la PCR, presentan ventajas como su gran especificidad, el hecho de ser rápidos y de fácil realización, y que en muchos casos ni siquiera es necesario un tratamiento previo de la muestra, lo que facilita su incorporación a las plantas de elaboración. En este trabajo se describen algunos de los métodos disponibles en la actualidad para la detección de microorganismos productores de aminas biógenas, así como sus posibles aplicaciones.
BIOGENIC AMINES IN FOOD: MOLECULAR METHODS FOR THE DETECTION AND IDENTIFICATION OF BA-PRODUCING BACTERIA
ABSTRACT
Biogenic amines are low molecular weight nitrogenous compounds with biological activity that are formed by decarboxylation of certain amino acids. Biogenic amines are present in all living organisms, in which they have important physiological functions. However, due to the metabolism of certain microorganisms, these compounds can accumulate in food at high concentrations, constituting a health risk for consumers. The presence of biogenic amine-producing microorganisms is the indispensable condition for the presence and further accumulation of biogenic amines in food. Because of this, several methods for the detection and identification of these biogenic amine-producing microorganisms have been developed. Among them, those based on culture independent techniques, such as the PCR, have several advantages such as their specificity, they are methods that are fast and easy to perform, and in many cases there is no need for pre-processing of food material which facilitates their incorporation at production plants. In this work, some available methods for the detection of biogenic amine-producing microorganisms are described, as well as their potential applications.
Recibido: 26-02-2019; Aceptado: 29-10-2019.
Cómo citar este artículo/Citation: Río, B. del; Redruello, B.; Fernández, M.; Ladero, V. y Álvarez, M.A. (2020). Aminas biógenas en alimentos: métodos moleculares para la detección e identificación de bacterias productoras. Arbor, 196 (795): a545. https://doi.org/10.3989/arbor.2020.795n1009
PALABRAS CLAVE: aminas biógenas; tiramina; histamina; putrescina; alimentos; PCR; métodos moleculares.
KEYWORDS: biogenic amines; tyramine; histamine; putrescine; food; PCR; molecular methods.
Copyright: © 2020 CSIC. Este es un artículo de acceso abierto distribuido bajo los términos de la licencia de uso y distribución Creative Commons Reconocimiento 4.0 Internacional (CC BY 4.0).
CONTENIDOS
LAS AMINAS BIÓGENAS Top
Las aminas biógenas (AB) son pequeños compuestos nitrogenados (< 200 Dalton), no proteicos, que contienen uno o más grupos amino y que se forman por descarboxilación enzimática de determinados aminoácidos. Las AB están presentes en todos los seres vivos, donde participan en procesos biológicos tan importantes como la replicación del ADN, la regulación del metabolismo, la respuesta inmune o la trasmisión del impulso nervioso (Ladero, Calles-Enríquez, Fernández y Álvarez, 2010Ladero, V., Calles-Enríquez, M., Fernández, M. y Álvarez, M. A. (2010). Toxicological effects of dietary biogenic amines. Current Nutrition and Food Science, 6 (2), pp. 145-156. https://doi.org/10.2174/157340110791233256.). Sin embargo, debido al metabolismo de algunos microorganismos, en algunos alimentos se pueden acumular AB en concentraciones tan elevadas que su ingesta puede provocar intoxicaciones (Ladero, Calles-Enríquez et al., 2010Ladero, V., Calles-Enríquez, M., Fernández, M. y Álvarez, M. A. (2010). Toxicological effects of dietary biogenic amines. Current Nutrition and Food Science, 6 (2), pp. 145-156. https://doi.org/10.2174/157340110791233256.).
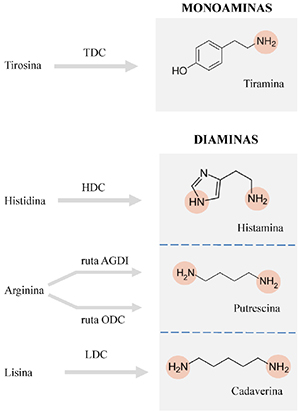
Las AB más frecuentes en alimentos son la tiramina (monoamina), la histamina, la putrescina y la cadaverina (diaminas) (Silla Santos, 1996Silla Santos, M. H. (1996). Biogenic amines: their importance in foods. International Journal of Food Microbiology, 29 (2-3), pp. 213-231. https://doi.org/10.1016/0168-1605(95)00032-1.). Estas AB se acumulan en los alimentos debido a la actividad de ciertas enzimas de origen microbiano que descarboxilan el correspondiente aminoácido precursor; así, la tirosina es el precursor de la tiramina, la histidina de la histamina y la lisina de la cadaverina. La putrescina se puede sintetizar por dos rutas alternativas: bien mediante descarboxilación de ornitina, o bien a partir de arginina en dos reacciones sucesivas de descarboxilación y desaminación (véase figura 1). Tradicionalmente se han incluido las poliaminas espermina y espermidina en el grupo de las AB, pero en su biosíntesis no participan descarboxilasas, sino que se realiza mediante reacciones sucesivas de condensación de la putrescina. Teniendo también en cuenta que sus funciones biológicas son diferentes, considerarlas AB es discutible (Bardocz, 1999Bardocz, S. (1999). Role of biogenic amines-summing up or what is it we do not know?. En: Bardocz, S., Koninkx, J., Grillo, M. y White, A. (eds.). Biogenically active amines in food (vol. III), pp. 1-4. Disponible en https://op.europa.eu/en/publication-detail/-/publication/ed5a2fd4-32fd-48f7-8175-18a3d28e3b5e/language-en/format-PDF/source-search.).
|
Figura 1. Estructura química y biosíntesis de las principales aminas biógenas que se pueden acumular en alimentos. Los grupos amino se representan mediante círculos coloreados. TDC, tirosina descarboxilasa; HDC, histidina descarboxilasa; AGDI, Agmatina deiminasa; ODC, ornitina descarboxilasa; LDC, lisina descarboxilasa
[Descargar tamaño completo]
| |
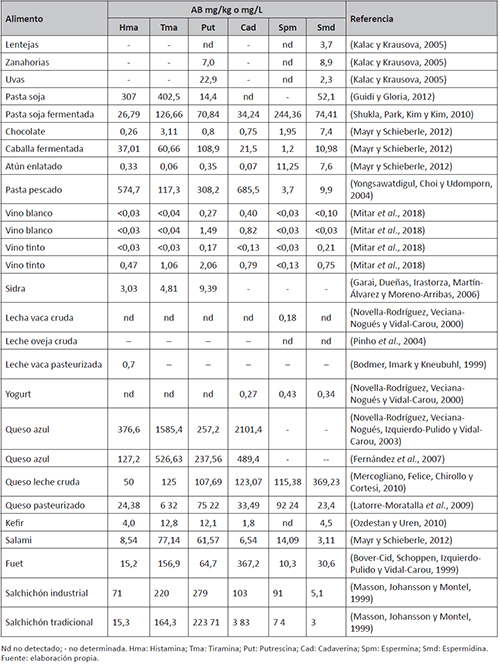
En la fruta, verdura, carne o pescado frescos, la concentración de AB suele ser baja y se atribuye al metabolismo normal de las células de sus tejidos (véase tabla 1). Sin embargo, su concentración puede aumentar considerablemente si las condiciones higiénicas durante la manipulación o almacenamiento de estos alimentos favorecen el crecimiento de microorganismos productores de AB. Por lo tanto, la presencia de AB es un indicador de la frescura y calidad higiénica de estos alimentos (Ladero, Calles-Enríquez et al., 2010Ladero, V., Calles-Enríquez, M., Fernández, M. y Álvarez, M. A. (2010). Toxicological effects of dietary biogenic amines. Current Nutrition and Food Science, 6 (2), pp. 145-156. https://doi.org/10.2174/157340110791233256.). Sin embargo, en bebidas y alimentos fermentados la biosíntesis de AB se debe mayoritariamente a la acción de microorganismos Gram positivos, pertenecientes al grupo de las bacterias del ácido láctico (BAL), que pueden estar presentes en la materia prima e incluso ser incluidos como iniciadores de la fermentación, sin descartar que también puedan ser microorganismos contaminantes (Álvarez y Moreno-Arribas, 2014Álvarez, M. A. y Moreno-Arribas, M. V. (2014). The problem of biogenic amines in fermented foods and the use of potential biogenic amine-degrading microorganisms as a solution. Trends in Food Science & Technology, 39 (2), pp. 146-155. https://doi.org/10.1016/j.tifs.2014.07.007.; Linares, Cruz Martín, Ladero, Álvarez y Fernández, 2011Linares, D. M., Cruz Martín, M., Ladero, V., Álvarez, M. A. y Fernández, M. (2011). Biogenic amines in dairy products. Critical Reviews in Food Science and Nutrition, 51 (7), pp. 691-703. https://doi.org/10.1080/10408398.2011.582813.; Novella-Rodríguez, Veciana-Nogués, Roig-Sagués, Trujillo-Mesa y Vidal-Carou, 2004Novella-Rodríguez, S. N., Veciana-Nogués, M. T., Roig-Sagués, A. X., Trujillo-Mesa, A .J. y Vidal-Carou, M. C. (2004). Evaluation of biogenic amines and microbial counts throughout the ripening of goat cheeses from pasteurized and raw milk. Journal of Dairy Research, 71 (2), pp. 245-252. https://doi.org/10.1017/S0022029904000147.). En estos alimentos, las AB suelen encontrarse más frecuentemente y en concentraciones más altas como consecuencia de las actividades descarboxilasa de estos microorganismos, pudiendo alcanzar valores que constituyen un riesgo para los consumidores (Fernández, Linares, Río, Ladero y Álvarez, 2007Fernández, M., Linares, D. M., Río, B. del, Ladero, V. y Álvarez, M. A. (2007). HPLC quantification of biogenic amines in cheeses: correlation with PCR-detection of tyramine-producing microorganisms. Journal of Dairy Research, 74 (3), pp. 276-282. https://doi.org/10.1017/S0022029907002488.; EFSA, 2011EFSA Panel on BiologicalHazards (BIOHAZ) (2011). Scientific Opinion on risk based control of biogenic amine formation in fermented foods. EFSA Journal, 9 (10), pp. 2393-2486.). Sin embargo, debido a que los principales productores son BAL, la presencia de AB en productos fermentados no puede considerarse como un indicador de su calidad higiénica y microbiológica.
|
Tabla 1. Contenido en aminas biógenas en diversos tipos de alimentos. Los valores se indican en mg/kg para alimentos sólidos o en mg/L para alimentos líquidos.
[Descargar tamaño completo]
| |
TOXICIDAD Top
Las AB ingeridas en la dieta son eficientemente metabolizadas en la mucosa intestinal mediante reacciones catalizadas por amino oxidasas específicas. Sin embargo, la ingesta de alimentos con elevadas concentraciones de AB puede saturar este sistema de detoxificación, no siendo suficiente para eliminar las AB presentes en el intestino, las cuales pueden pasar al torrente sanguíneo provocando diversos efectos tóxicos. Además, el sistema de detoxificación puede tener reducida su eficacia debido a particularidades individuales de origen genético o derivadas del consumo de sustancias inhibidoras de las amino oxidasas como el alcohol, el tabaco o determinados fármacos antidepresivos. En estos casos la cantidad de AB ingerida capaz de provocar reacciones adversas es menor (Blackwell, 1963Blackwell, B. (1963). Hypertensive crisis due to monoamine-oxidase inhibitors. Lancet, 282 (7313), pp. 849-851. https://doi.org/10.1016/S0140-6736(63)92743-0.; Ladero, Calles-Enríquez et al., 2010Ladero, V., Calles-Enríquez, M., Fernández, M. y Álvarez, M. A. (2010). Toxicological effects of dietary biogenic amines. Current Nutrition and Food Science, 6 (2), pp. 145-156. https://doi.org/10.2174/157340110791233256.). Las AB no eliminadas pueden provocar alteraciones gastrointestinales (náuseas, vómitos, diarrea, dolores abdominales), hemodinámicas (hipotensión, hipertensión) y cutáneas (comezón, urticaria, edema, inflamaciones).
Debido a estos efectos tóxicos, sería necesario establecer unos límites máximos de la concentración de AB en los alimentos. Sin embargo, la European Food Safety Authority (EFSA) ha reconocido que uno de los problemas a los que se enfrenta a la hora de hacer recomendaciones para regular la presencia de AB en alimentos es la falta de suficientes estudios científicos sobre su toxicidad (EFSA, 2011EFSA Panel on BiologicalHazards (BIOHAZ) (2011). Scientific Opinion on risk based control of biogenic amine formation in fermented foods. EFSA Journal, 9 (10), pp. 2393-2486.). Además, la absorción y la toxicidad de una AB puede verse alterada por la presencia de otra amina, produciéndose sinergias que dificultan aún más establecer un límite (Silla Santos, 1996Silla Santos, M. H. (1996). Biogenic amines: their importance in foods. International Journal of Food Microbiology, 29 (2-3), pp. 213-231. https://doi.org/10.1016/0168-1605(95)00032-1.). Encaminados hacia ese objetivo, nuestro grupo de investigación ha desarrollado un sistema in vitro de evaluación de la citotoxicidad de las AB, enfrentándolas a cultivos celulares intestinales de origen humano (Linares et al., 2016Linares, D. M., Río, B. del, Redruello, B., Ladero, V., Martín, M. C., Fernández, M., Ruas-Mediedo, P. y Álvarez, M. A. (2016). Comparative analysis of the in vitro cytotoxicity of the dietary biogenic amines tyramine and histamine. Food Chemistry, 197, pp. 658-663. https://doi.org/10.1016/j.foodchem.2015.11.013.). Con este sistema hemos determinado la citotoxicidad y el modo de acción de la tiramina, la histamina, la putrescina y la cadaverina a concentraciones en las que se pueden encontrar en los alimentos (Linares et al., 2016Linares, D. M., Río, B. del, Redruello, B., Ladero, V., Martín, M. C., Fernández, M., Ruas-Mediedo, P. y Álvarez, M. A. (2016). Comparative analysis of the in vitro cytotoxicity of the dietary biogenic amines tyramine and histamine. Food Chemistry, 197, pp. 658-663. https://doi.org/10.1016/j.foodchem.2015.11.013.; Río et al., 2019Río, B. del, Redruello, B., Linares, D. M., Ladero, V., Ruas-Madiedo, P., Fernández, M., Martin, M. C. y Álvarez, M. A. (2019). The biogenic amines putrescine and cadaverine show in vitro cytotoxicity at concentrations that can be found in foods. Scientific Reports, 9 (1), e120. https://doi.org/10.1038/s41598-018-36239-w.). La tiramina resultó ser la AB más citotóxica seguida de la histamina, y además hemos demostrado que la tiramina y la histamina tienen un efecto citotóxico sinérgico (Río et al., 2017Río, B. del, Redruello, B., Linares, D. M., Ladero, V., Fernández, M., Martín, M. C., Ruas-Madiedo, P. y Álvarez, M. A. (2017). The dietary biogenic amines tyramine and histamine show synergistic toxicity towards intestinal cells in culture. Food Chemistry, 218, pp. 249-255. https://doi.org/10.1016/j.foodchem.2016.09.046.).
A pesar de sus efectos tóxicos y de las altas concentraciones que pueden alcanzar las AB en algunos alimentos fermentados, solo se ha regulado legalmente el contenido máximo de histamina y únicamente para pescados y productos derivados del pescado; así, la legislación europea (Directiva 91/439/CEE) ha establecido un límite máximo de 100 mg/kg para pescado crudo y 200 mg/kg para pescado salado, mientras que la legislación estadounidense lo ha establecido en 50 mg/kg (véase Scombro toxin (histamine) formation). Para otros alimentos aún no se han establecido límites máximos de histamina, solo existen recomendaciones. Por ejemplo, en el caso de la histamina las concentraciones superiores a 400 mg/kg se consideran peligrosas para la salud (Taylor y World Health Organization, 1985Taylor, S. L. y World Health Organization (1985). Histamine poisoning associated with fish, cheese and other foods. World Health Organization. Report VPH/FOS/85.1. [En línea]. Disponible en https://apps.who.int/iris/handle/10665/66407.). Respecto al resto de AB, no existe un consenso claro, y diversos autores recomiendan no exceder 200-500 mg/kg en el caso de la tiramina o entre 800-1000 mg/kg para la presencia total de aminas (Silla Santos, 1996Silla Santos, M. H. (1996). Biogenic amines: their importance in foods. International Journal of Food Microbiology, 29 (2-3), pp. 213-231. https://doi.org/10.1016/0168-1605(95)00032-1.; ten Brink, Damink, Joosten y Tveld, 1990ten Brink, B., Damink, C., Joosten, H. M. y Tveld, J. H. (1990). Occurrence and formation of biologically-active amines in foods. International Journal of Food Microbiology, 11 (1), pp. 73-84. https://doi.org/10.1016/0168-1605(90)90040-C.).
MICROORGANISMOS PRODUCTORES DE AMINAS BIÓGENAS Top
La acumulación de AB en los alimentos requiere como condición indispensable la presencia de los microorganismos productores. Se han descrito una gran variedad de bacterias presentes en alimentos con la capacidad de producir AB, incluyendo tanto bacterias Gram positivas como Gram negativas (Gardini, Ozogul, Suzzi, Tabanelli y Ozogul, 2016Gardini, F., Ozogul, Y., Suzzi, G., Tabanelli, G. y Ozogul, F. (2016). Technological factors affecting biogenic amine content in foods: a review. Frontiers in Microbiology, 7, 1218. https://doi.org/10.3389/fmicb.2016.01218.). Dentro de las Gram negativas, el mayor número de bacterias productoras de AB pertenecen a la familia Enterobacteriaceae, las cuales han sido asociadas con la producción de histamina, principalmente en pescados, y putrescina y cadaverina en carnes, derivados cárnicos y productos lácteos. Las bacterias productoras de esta familia pertenecen a los géneros Morganella, Hafnia, Klebsiella, Raoutella, Escherichia, Enterobacter y Serratia (Geornaras, Dykes y von Holy, 1995Geornaras, I., Dykes, G. A. y von Holy, A. (1995). Biogenic amine formation by poultry-associated spoilage and pathogenic bacteria. Letters in Applied Microbiology, 21 (3), pp. 164-166. https://doi.org/10.1111/j.1472-765X.1995.tb01032.x.; Kim et al., 2001Kim, S. H., Field, K. G., Morrissey, M. T., Price, R. J., Wei, C. I. y An, H. (2001). Source and identification of histamine-producing bacteria from fresh and temperature-abused albacore. Journal of Food Protection, 64 (7), pp. 1035-1044. https://doi.org/10.4315/0362-028X-64.7.1035.). En la familia Pseudomonales se han identificado cepas de diferentes especies de Pseudomonas productoras de histamina y putrescina (Fernández-No, Bohme, Calo-Mata y Barros-Velázquez, 2011Fernández-No, I. C., Bohme, K., Calo-Mata, P. y Barros-Velázquez, J. (2011). Characterisation of histamine-producing bacteria from farmed blackspot seabream (Pagellus bogaraveo) and turbot (Psetta maxima). International Journal of Food Microbiology, 151 (2), pp. 182-189. https://doi.org/10.1016/j.ijfoodmicro.2011.08.024.). Así mismo, en la familia Vibronoceae se ha identificado Photobacterium phosphoreum como productor de histamina (Jorgensen, Huss y Dalgaard, 2000Jorgensen, L. V., Huss, H. H. y Dalgaard, P. (2000). The effect of biogenic amine production by single bacterial cultures and metabiosis on cold-smoked salmon. Journal of Applied Microbiology, 89 (6), pp. 920-934. https://doi.org/10.1046/j.1365-2672.2000.01196.x.).
La lista de bacterias Gram positivas identificadas como productoras de AB en alimentos incluye BAL de los géneros Lactococcus, productores de putrescina; Enterococcus, productores de putrescina y tiramina; Lactobacillus, productores de histamina, putrescina y tiramina, Oenococcus y Pediococcus, productores de histamina, y Streptococcus, productores de tiramina e histamina. Dentro del género Staphylococcus se han identificado bacterias productoras de histamina, putrescina, cadaverina y tiramina (Cachaldora, Fonseca, Franco y Carballo, 2013Cachaldora, A., Fonseca, S., Franco, I. y Carballo, J. (2013). Technological and safety characteristics of Staphylococcaceae isolated from Spanish traditional dry-cured sausages. Food Microbiology, 33 (1), pp. 61-68. https://doi.org/10.1016/j.fm.2012.08.013.), siendo las especies Staphylococcus saprophyticus, Staphylococcus simulans y Staphylococcus xylosus las que presentan un mayor porcentaje de cepas productoras (Martín et al., 2006Martín, B., Garriga, M., Hugas, M., Bover-Cid, S., Veciana-Nogués, M. T. y Aymerich, T. (2006). Molecular, technological and safety characterization of Gram-positive catalase-positive cocci from slightly fermented sausages. International Journal of Food Microbiology, 107 (2), pp. 148-158. https://doi.org/10.1016/j.ijfoodmicro.2005.08.024.). En el caso del género Bacillus existe muy poca información sobre la actividad aminogénica de sus miembros, aunque Bermúdez, Lorenzo, Fonseca, Franco y Carballo (2012Bermúdez, R., Lorenzo, J. M., Fonseca, S., Franco, I. y Carballo, J. (2012). Strains of Staphylococcus and Bacillus isolated from traditional sausages as producers of biogenic amines. Frontiers in Microbiology, 3, 151. https://doi.org/10.3389/fmicb.2012.00151.) identificaron algunas cepas de Bacillus subtilis y Bacillus amyloliquefacis aisladas de carne que presentaban actividad, lisina, tirosina y ornitina descarboxilasa.
Durante muchos años se propuso que la capacidad para producir aminas biógenas debía ser considerada como una característica de cepa y no de especie. De hecho, en especies como Oenococcus oeni, Streptococcus thermophilus, Lactobacillus parabuchneri o Lactobacillus brevis se ha demostrado que la producción de aminas biógenas es una característica de cepa, ya que no está presente en todas las cepas de la especie, y además los genes implicados en su síntesis están asociados a elementos genéticos móviles, por lo que se piensa que esta capacidad ha sido adquirida mediante transferencia genética horizontal (Calles-Enríquez et al., 2010Calles-Enríquez, M., Eriksen, B. H., Andersen, P. S., Rattray, F. P., Johansen, A. H., Fernández, M., Ladero, V. y Álvarez, M. A. (2010). Sequencing and transcriptional analysis of the Streptococcus thermophilus histamine biosynthesis gene cluster: factors that affect differential hdcA expression. Applied and Environmental Microbiology, 76 (18), pp. 6231-6238. https://doi.org/10.1128/Aem.00827-10.; Marcobal, Rivas, Moreno-Arribas, y Muñoz, 2006Marcobal, A., Rivas, B. de las, Moreno-Arribas, M. V. y Muñoz, R. (2006). Evidence for horizontal gene transfer as origin of putrescine production in Oenococcus oeni RM83. Applied and Environmental Microbiology, 72 (12), pp. 7954-7958. https://doi.org/10.1128/Aem.01213-06.; Romano, Ladero, Álvarez y Lucas, 2014Romano, A., Ladero, V., Álvarez, M. A. y Lucas, P. M. (2014). Putrescine production via the ornithine decarboxylation pathway improves the acid stress survival of Lactobacillus brevis and is part of a horizontally transferred acid resistance locus. International Journal of Food Microbiology, 175, pp. 14-19. https://doi.org/10.1016/j.ijfoodmicro.2014.01.009.; Wuthrich et al., 2017Wuthrich, D., Berthoud, H., Wechsler, D., Eugster, E., Irmler, S. y Bruggmann, R. (2017). The histidine decarboxylase gene cluster of Lactobacillus parabuchneri was gained by horizontal gene transfer and is mobile within the species. Frontiers in Microbiology, 8, 218. https://doi.org/10.3389/fmicb.2017.00218.). Sin embargo, al aumentar el número de trabajos encaminados a identificar cepas productoras de AB, hoy en día es posible afirmar que en algunas especies, como es el caso de Enterococcus faecalis, Enterococcus faecium, o Enterococcus durans, la capacidad para sintetizar AB puede ser considerada una característica de la especie (Ladero, Fernández et al. 2012Ladero, V., Fernández, M., Calles-Enríquez, M., Sánchez-Llana, E., Cañedo, E., Martín, M. C. y Álvarez, M. A. (2012). Is the production of the biogenic amines tyramine and putrescine a species-level trait in enterococci?. Food Microbiology, 30 (1), pp. 132-138. https://doi.org/10.1016/J.Fm.2011.12.016.). Un caso particular es el de la especie Lactococcus lactis, la más utilizada como cultivo iniciador en quesos, en la que la capacidad de producir putrescina es considerada una característica de especie (Ladero, Rattray et al., 2011Ladero, V., Rattray, F. P., Mayo, B., Martín, M. C., Fernández, M. y Álvarez, M. A. (2011). Sequencing and transcriptional analysis of the biosynthesis gene cluster of putrescine-producing Lactococcus lactis. Applied and Environmental Microbiology, 77 (18), pp. 6409-6418. https://doi.org/10.1128/Aem.05507-11.). Sin embargo, se ha visto que en el proceso de domesticación y adaptación al entorno lácteo se han seleccionado cepas que han perdido esta capacidad (Ladero, Rattray et al., 2011Ladero, V., Rattray, F. P., Mayo, B., Martín, M. C., Fernández, M. y Álvarez, M. A. (2011). Sequencing and transcriptional analysis of the biosynthesis gene cluster of putrescine-producing Lactococcus lactis. Applied and Environmental Microbiology, 77 (18), pp. 6409-6418. https://doi.org/10.1128/Aem.05507-11.), por lo que aun siendo una característica de especie, no todas las cepas de Lactococcus lactis presentan la capacidad de producir putrescina.
El hecho de que algunas cepas de especies utilizadas como cultivos iniciadores, como Lactococcus lactis, Oenococcus oeni o Streptococcus thermophilus, presenten la capacidad de producir AB hace que disponer de métodos adecuados para detectar estas cepas, y por lo tanto no incluirlas como cultivos iniciadores, sea de vital importancia para prevenir la acumulación de AB en alimentos fermentados.
GENÉTICA DE LA PRODUCCIÓN DE AMINAS BIÓGENAS Top
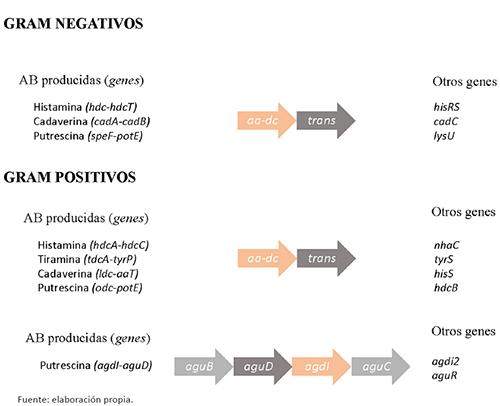
Las AB se forman en el interior celular por la descarboxilación enzimática de un aminoácido y posteriormente son secretadas al exterior mediante un transportador de tipo antiporter, el cual incorpora una molécula del aminoácido sustrato por cada molécula de AB generada que expulsa. Por lo tanto, se necesitan al menos dos proteínas, la aminoacil-descarboxilasa y el transportador, que son codificadas por dos genes distintos (véase figura 2). En bacterias Gram negativas, esta organización es la más extendida. Sin embargo, en bacterias Gram positivas estos dos genes suelen estar acompañados de otros genes (figura 2) que codifican diversas funciones accesorias relacionadas con la maduración o estabilidad del enzima, como es el caso de hdcB en el cluster de producción de histamina en Streptococcus thermophilus (Trip, Mulder, Rattray y Lolkema, 2011Trip, H., Mulder, N. L., Rattray, F. P. y Lolkema, J. S. (2011). HdcB, a novel enzyme catalysing maturation of pyruvoyl-dependent histidine decarboxylase. Molecular Microbiology, 79 (4), pp. 861-871. https://doi.org/10.1111/j.1365-2958.2010.07492.x.), intercambiadores de iones como nhaC en el cluster de producción de tiramina en Enterococcus faecalis (Pérez et al., 2015Pérez, M., Calles-Enríquez, M., Nes, I., Martín, M. C., Fernández, M., Ladero, V. y Álvarez, M. A. (2015). Tyramine biosynthesis is transcriptionally induced at low pH and improves the fitness of Enterococcus faecalis in acidic environments. Applied Microbiology and Biotechnology, 99 (8), pp. 3547-3558. https://doi.org/10.1007/s00253-014-6301-7.), o enzimas con similitud a aminoacil-tRNA sintetasas en Lactobacillus parabuchneri (hisS) o Enterococcus durans (tyrS) con función desconocida (Linares et al., 2012Linares, D. M., Fernández, M., Río, B. del, Ladero, V., Martín, M. C. y Álvarez, M. A. (2012). The tyrosyl-tRNA synthetase like gene located in the tyramine biosynthesis cluster of Enterococcus durans is transcriptionally regulated by tyrosine concentration and extracellular pH. BMC Microbiology, 12 (23). https://doi.org/10.1186/1471-2180-12-23.; Martín, Fernández, Linares y Álvarez, 2005Martín, M. C., Fernández, M., Linares, D. M. y Álvarez, M. A. (2005). Sequencing, characterization and transcriptional analysis of the histidine decarboxylase operon of Lactobacillus buchneri. Microbiology-Sgm, 151, pp. 1219-1228. https://doi.org/10.1099/mic.0.27459-0.). Un caso más complejo es la producción de putrescina a partir de la arginina. La putrescina se forma tras dos reacciones consecutivas de descarboxilación y desaminación de la arginina y dependiendo del orden de estas dos reacciones se han descrito dos posibles rutas. La ruta ODC, en la que la arginina es desaminada dando ornitina y posteriormente descarboxilada dando putrescina, y la ruta AGDI, en la que la arginina se descarboxila dando agmatina que posteriormente es desaminada mediante varias reacciones enzimáticas para formar putrescina (véase figura 2).
|
Figura 2. Representación esquemática de los genes implicados en la síntesis de las principales AB. Se indican los nombres más comunes de los genes de cada una de las aminoácil descarboxilasas y sus transportadores. Asimismo, se indican los nombres de otros genes accesorios que acompañan a éstos. En el caso de la ruta de la agmatina deiminasa, se muestran en la figura el resto de genes catalíticos implicados en la reacción de desaminación de la agmatina. (ver más detalles en el texto)
[Descargar tamaño completo]
| |
En los últimos años se han identificado y secuenciado una gran variedad de genes implicados en la producción de AB de un gran número de especies tanto Gram positivas como Gram negativas. El análisis de la secuencia de nucleótidos de estos genes ha permitido ver que tienen una serie de regiones conservadas que flanquean regiones variables, y este hecho permite que se puedan diseñar sistemas de detección independientes de cultivo para la detección e identificación de bacterias productoras de AB, como veremos en detalle más adelante.
MÉTODOS DE DETECCIÓN DE MICROORGANISMOS PRODUCTORES DE AMINAS BIÓGENAS Top
Hasta la fecha se han diseñado distintos métodos que permiten la detección de bacterias lácticas productoras de AB tanto en cultivos iniciadores como en diferentes matrices alimentarias. Estos métodos se clasifican en a) métodos dependientes de cultivo, que se basan en el crecimiento del microorganismo en medios de cultivo diferenciales que ponen de manifiesto la actividad aminoácido descarboxilasa correspondiente, y b) métodos independientes de cultivo, que no requieren el crecimiento del microorganismo, y que se basan en técnicas moleculares, alguna de las cuales permiten además de detectar, cuantificar e incluso identificar las especies bacterianas potencialmente productoras de AB.
Métodos dependientes de cultivo
Los métodos utilizados inicialmente para detectar BAL productoras de AB se basan en el aislamiento de los microorganismos del alimento o del cultivo iniciador y en su posterior crecimiento en un medio de cultivo sólido diferencial suplementado con el aminoácido sustrato y un indicador de pH (por ejemplo, el púrpura de bromocresol) que vira de color cuando el pH del medio se alcaliniza (Bover-Cid y Holzapfel, 1999Bover-Cid, S. y Holzapfel, W. H. (1999). Improved screening procedure for biogenic amine production by lactic acid bacteria. International Journal of Food Microbiology, 53 (1), pp. 33-41. https://doi.org/10.1016/S0168-1605(99)00152-X.; Joosten y Northolt, 1989Joosten, H. M. y Northolt, M. D. (1989). Detection, growth, and amine-producing capacity of lactobacilli in cheese. Applied and Environmental Microbiology, 55 (9), pp. 2356-2359.; Maijala, 1993Maijala, R. L. (1993). Formation of histamine and tyramine by some lactic acid bacteria in MRS-broth and modified decarboxylation agar. Letters in Applied Microbiology, 17 (1), pp. 40-43. https://doi.org/10.1111/j.1472-765X.1993.tb01431.x.). Si la bacteria tiene actividad aminoácido descarboxilasa y sintetiza la AB correspondiente, el pH del medio aumentaría debido a que la AB producida es más básica que el aminoácido sustrato y se produciría un halo de diferente color alrededor de la colonia productora. Utilizando este tipo de medios diferenciales se han podido detectar cepas de BAL aisladas de cultivos iniciadores o de distintos alimentos capaces de producir diferentes AB (Enterococcus faecalis, Enterococcus faecium, Enterococcus durans, Carnobacterium divergens, Carnobacterium piscicola, Lactobacillus curvatus, o Lactobacillus brevis entre otras especies (Bover-Cid, Hugas, Izquierdo-Pulido y Vidal-Carou, 2001Bover-Cid, S., Hugas, M., Izquierdo-Pulido, M. y Vidal-Carou, M C. (2001). Amino acid-decarboxylase activity of bacteria isolated from fermented pork sausages. International Journal of Food Microbiology, 66 (3), pp. 185-189. https://doi.org/10.1016/S0168-1605(00)00526-2.; Joosten y Northolt, 1989Joosten, H. M. y Northolt, M. D. (1989). Detection, growth, and amine-producing capacity of lactobacilli in cheese. Applied and Environmental Microbiology, 55 (9), pp. 2356-2359.). Estos medios han sido especialmente eficaces para detectar BAL productoras de tiramina (Ladero, Martínez, Martín, Fernández y Álvarez, 2010Ladero, V., Martínez, N., Martín, M. C., Fernández, M. y Álvarez, M. A. (2010). qPCR for quantitative detection of tyramine-producing bacteria in dairy products. Food Research International, 43 (1), pp. 289-295. https://doi.org/10.1016/j.foodres.2009.10.007.), ya que este aminoácido es muy insoluble y forma un precipitado opaco en el medio de descarboxilación sólido; el medio se torna transparente alrededor de aquellas cepas productoras de tiramina, haciéndolas fácilmente identificables. Sin embargo, los medios de descarboxilación sólidos no han demostrado ser especialmente eficaces para detectar BAL productoras de histamina, a pesar de que se ha descrito el aislamiento de algunas de estas cepas de alimentos, como Lactobacillus buchneri (Joosten y Northolt, 1989Joosten, H. M. y Northolt, M. D. (1989). Detection, growth, and amine-producing capacity of lactobacilli in cheese. Applied and Environmental Microbiology, 55 (9), pp. 2356-2359.) actualmente identificado como Lactobacillus parabuchneri (Díaz, Río et al., 2016Díaz, M., Río, B. del, Sánchez-Llana, E., Ladero, V., Redruello, B., Fernández, M., Martín, M. C. y Álvarez, M. A. (2016). Histamine-producing Lactobacillus parabuchneri strains isolated from grated cheese can form biofilms on stainless steel. Food Microbiology, 59, pp. 85-91. https://doi.org/10.1016/j.fm.2016.05.012.; Díaz, Ladero, Río et al., 2016Díaz, M., Ladero, V., Río, B. del, Redruello, B., Fernández, M., Martín, M. C. y Álvarez, M. A. (2016). Biofilm-forming capacity in biogenic amine-producing bacteria isolated from dairy products. Frontiers in Microbiology, 7, 591. https://doi.org/10.3389/fmicb.2016.00591.). Esto se debe a que el ácido láctico producido por estas bacterias contrarresta la alcalinidad de la histamina acumulada y no se produce el viraje de color del indicador de pH. Sin embargo, el uso de medios de descarboxilación líquidos con baja concentración de glucosa ha permitido detectar más cepas productoras de histamina aisladas de alimentos como el queso (Lactobacillus vaginalis (Díaz et al., 2015Díaz, M., Río, B. del, Ladero, V., Redruello, B., Fernández, M., Martín, M. C. y Álvarez, M. A. (2015). Isolation and typification of histamine-producing Lactobacillus vaginalis strains from cheese. International Journal of Food Microbiology, 215, pp. 117-123. https://doi.org/10.1016/j.ijfoodmicro.2015.08.026.), Lactobacillus parabuchneri (Díaz, Ladero, Río et al., 2016Díaz, M., Ladero, V., Río, B. del, Redruello, B., Fernández, M., Martín, M. C. y Álvarez, M. A. (2016). Biofilm-forming capacity in biogenic amine-producing bacteria isolated from dairy products. Frontiers in Microbiology, 7, 591. https://doi.org/10.3389/fmicb.2016.00591.)). Estos métodos se suelen combinar con métodos analíticos (tales como electroforesis en capa fina (Thin Layer Chromatography, TLC (Latorre-Moratalla, Bover-Cid, Veciana-Nogués y Vidal-Carou, 2009Latorre-Moratalla, M. L., Bover-Cid, S., Veciana-Nogués, T. y Vidal-Carou, M. C. (2009). Thin-layer chromatography for the identification and semi-quantification of biogenic amines produced by bacteria. Journal of Chromatography A, 1216 (18), pp. 4128-4132. https://doi.org/10.1016/j.chroma.2009.02.045.), cromatografía líquida de alta resolución (High Performance Liquid Chromatography, HPLC (Fernández et al., 2007Fernández, M., Linares, D. M., Río, B. del, Ladero, V. y Álvarez, M. A. (2007). HPLC quantification of biogenic amines in cheeses: correlation with PCR-detection of tyramine-producing microorganisms. Journal of Dairy Research, 74 (3), pp. 276-282. https://doi.org/10.1017/S0022029907002488.) o cromatografía líquida de ultra alta resolución (Ultra High Performance Liquid Chromatography, UHPLC (Redruello et al., 2013Redruello, B., Ladero, V., Cuesta, I., Álvarez-Buylla, J. R., Martín, M. C., Fernández, M. y Álvarez, M. A. (2013). A fast, reliable, ultra high performance liquid chromatography method for the simultaneous determination of amino acids, biogenic amines and ammonium ions in cheese, using diethyl ethoxymethylenemalonate as a derivatising agent. Food Chemistry, 139 (1-4), pp. 1029-1035. https://doi.org/10.1016/j.foodchem.2013.01.071.; Redruello et al., 2016Redruello, B., Ladero, V., Río, B. del, Fernández, M., Martín, M. C. y Álvarez, M. A. (2016). Data on recovery of 21 amino acids, 9 biogenic amines and ammonium ions after spiking four different beers with five concentrations of these analytes. Data in Brief, 9, pp. 398-400. https://doi.org/10.1016/j.dib.2016.09.011.)) diseñados para detectar y cuantificar la presencia de la AB en matrices alimentarias, ya que permiten confirmar la presencia de la AB en el medio de cultivo suplementado con el aminoácido correspondiente en el que se ha crecido la bacteria aislada.
Sin embargo, los métodos dependientes de cultivo tienen una serie de desventajas. En primer lugar, requieren el aislamiento de la bacteria del alimento, para lo que muy frecuentemente es necesario hacer cultivos de enriquecimiento que permitan aumentar su proporción en el cultivo, y su posterior crecimiento. Este proceso largo y tedioso no siempre resulta eficaz, ya que puede haber bacterias productoras que no sean capaces de crecer en estos medios de cultivo. Además, como estos medios no son selectivos, el aislamiento puede resultar muy difícil e incluso imposible en el caso de que la bacteria productora se encuentre en una proporción muy baja con respecto al número total de bacterias presentes en el alimento, el cual suele ser muy elevado en el caso concreto de los alimentos fermentados. También hay que tener en cuenta que pueden dar lugar a falsos positivos, si la bacteria produce algún compuesto básico distinto a las AB que sea capaz de alcalinizar el medio. Estos métodos, además, tienen baja sensibilidad y en función de la capacidad de producción y el estado fisiológico de las células ensayadas se pueden producir falsos negativos si la alcalinización debida a la producción de la AB se contrarresta con la acidez producida por el metabolismo fermentativo de las BAL. Además, aquellos que implican la detección de la producción mediante métodos cromatográficos requieren disponer de equipamiento sofisticado (Landete, Rivas, Marcobal y Muñoz, 2007Landete, J. M., Rivas, B. de las, Marcobal, A. y Muñoz, R. (2007). Molecular methods for the detection of biogenic amine-producing bacteria on foods. International Journal of Food Microbiology, 117 (3), pp. 258-269. https://doi.org/10.1016/j.ijfoodmicro.2007.05.001.). Por último, aunque no menos importante, estos métodos no son cuantitativos.
Una vez que se ha aislado el microorganismo productor de AB es necesario proceder a su identificación. Frecuentemente, las especies se han identificado de forma errónea, como es el caso de algunas cepas de Lactobacillus parabuchneri aisladas de queso y productoras de histamina que habían sido clasificadas como Lactobacillus buchneri, comentado anteriormente. Algunas de estas especies permanecen mal clasificadas tanto en publicaciones como en las bases de datos, lo que puede dar lugar a identificaciones incorrectas de otros aislados bacterianos y causar problemas en estudios futuros. Esto es debido a que en muchos casos la identificación se realizaba mediante métodos microbiológicos clásicos que se basan en criterios fenotípicos (microscópicos y macroscópicos) y en pruebas bioquímicas (determinación de actividad enzimática, metabolitos producidos, resistencia a antibióticos, etc.), en lugar de utilizar métodos genotípicos mucho más sólidos y precisos como los métodos moleculares basados en la secuenciación de genes con utilidad taxonómica (como gen del ARN ribosomal 16S (rna16S), Super óxido dismutasa (sod), subunidad β de la ARN polimerasa (rpoB), etc.).
Métodos independientes de cultivo
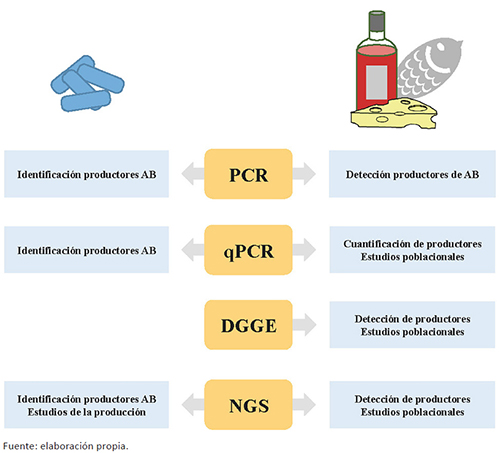
Frente a los métodos dependientes de cultivo, los métodos independientes de cultivo, especialmente los métodos moleculares basados en la reacción en cadena de la polimerasa (PCR), presentan varias ventajas destacables, como su gran especificidad y que son métodos rápidos y de fácil realización; de hecho, no es necesario disponer de una gran cantidad de ADN de alta pureza y en muchos casos ni siquiera es necesario un tratamiento previo de la muestra (Río et al., 2007Río, B. del, Binetti, A. G., Martín, M. C., Fernández, M., Magadan, A. H. y Álvarez, M. A. (2007). Multiplex PCR for the detection and identification of dairy bacteriophages in milk. Food Microbiology, 24 (1), pp. 75-81. https://doi.org/10.1016/j.fm.2006.03.001.; Martínez et al., 2011Martínez, N., Martín, M. C., Herrero, A., Fernández, M., Álvarez, M. A. y Ladero, V. (2011). qPCR as a powerful tool for microbial food spoilage quantification: Significance for food quality. Trends in Food Science & Technology, 22 (7), pp. 367-376. https://doi.org/10.1016/j.tifs.2011.04.004.). Por lo tanto, estos métodos pueden incorporarse a un laboratorio de alimentos e incluso a plantas de elaboración para el control de la calidad y seguridad microbiológica de los mismos. Además, dependiendo de la técnica utilizada y del diseño específico de la misma, las técnicas basadas en PCR permiten no solo determinar la capacidad de producción de aminas biógenas de una cepa, sino que pueden ser utilizadas para la cuantificación de bacterias productoras en los alimentos e incluso identificar las poblaciones presentes en los mismos (véase figura 3), lo que puede ayudar a tomar las medidas adecuadas para evitar la acumulación de aminas biógenas en cada tipo de alimento y estadio de elaboración (Ladero, Fernández y Álvarez, 2009Ladero, V., Fernández, M. y Álvarez, M. A. (2009). Effect of post-ripening processing on the histamine and histamine-producing bacteria contents of different cheeses. International Dairy Journal, 19 (12), pp. 759-762. https://doi.org/10.1016/j.idairyj.2009.05.010.; Landete, Rivas, Marcobal y Muñoz, 2011Landete, J. M., Rivas, B. de las, Marcobal, A. y Muñoz, R. (2011). PCR methods for the detection of biogenic amine-producing bacteria on wine. Annals of Microbiology, 61 (1), pp. 159-166. https://doi.org/10.1007/s13213-010-0068-6.; Martínez et al., 2011Martínez, N., Martín, M. C., Herrero, A., Fernández, M., Álvarez, M. A. y Ladero, V. (2011). qPCR as a powerful tool for microbial food spoilage quantification: Significance for food quality. Trends in Food Science & Technology, 22 (7), pp. 367-376. https://doi.org/10.1016/j.tifs.2011.04.004.).
|
Figura 3. Esquema de las diferentes aplicaciones de las técnicas moleculares de detección de genes implicados en la producción de AB tanto aplicadas a cultivos puros como a alimentos
[Descargar tamaño completo]
| |
Para el diseño de un método de detección bacteriana basado en PCR se necesita disponer de primers adecuados. Para poder diseñar los primers que nos permitan detectar específicamente los genes diana se hace necesario un conocimiento previo de estos genes y su comparación entre las bacterias portadoras. En el caso de los genes implicados en la producción de aminas biógenas, como hemos visto anteriormente, en los últimos años se ha realizado una descripción exhaustiva de estos genes en una gran variedad de géneros y especies, por lo que se dispone de suficiente información genética para su diseño. En algunos casos, si las secuencias de nucleótidos son muy divergentes, puede ser necesario el uso de más de una pareja de primers. Este es el caso de las bacterias con capacidad de producir histamina en alimentos, ya que pertenecen a grupos muy dispares, Gram negativas en pescados y derivados de los mismos y Gram positivas del grupo de las BAL en alimentos fermentados. En estos casos, se puede utilizar el método de PCR más adecuado según el grupo bacteriano que se intenta detectar o bien se realizan dos PCR, o se puede diseñar una PCR múltiple (Coton et al., 2010Coton, M., Romano, A., Spano, G., Ziegler, K., Vetrana, C., Desmarais, C., Lonvaud-Funel, A., Lucas, P. y Coton, E. (2010). Occurrence of biogenic amine-forming lactic acid bacteria in wine and cider. Food Microbiology, 27 (8), pp. 1078-1085. https://doi.org/10.1016/J.Fm.2010.07.012.; Rivas, Marcobal y Muñoz, 2005Rivas, B. de las, Marcobal, A. y Muñoz, R. (2005). Improved multiplex-PCR method for the simultaneous detection of food bacteria producing biogenic amines. FEMS Microbiology Letters, 244 (2), pp. 367-372. https://doi.org/10.1016/j.femsle.2005.02.012.).
Reacción en cadena de la polimerasa (PCR)
La PCR fue el primer desarrollo de técnicas independientes de cultivo para la detección de productores de AB. A partir de los genes implicados en la producción de AB cuyas secuencias estaban disponibles, se diseñaron primers específicos para la detección de productores de histamina (Le Jeune, Lonvaud-Funel, ten Brink, Hofstra y van der Vossen, 1995Le Jeune, C., Lonvaud-Funel, A., ten Brink, B., Hofstra, H. y van der Vossen, J. M. (1995). Development of a detection system for histidine decarboxylating lactic acid bacteria based on DNA probes, PCR and activity test. Journal of Appllied Bacteriology, 78 (3), pp. 316-326. https://doi.org/10.1111/j.1365-2672.1995.tb05032.x.) y tiramina (Lucas y Lonvaud-Funel, 2002Lucas, P. y Lonvaud-Funel, A. (2002). Purification and partial gene sequence of the tyrosine decarboxylase of Lactobacillus brevis IOEB 9809. FEMS Microbiology Letters, 211 (1), pp. 85-89. https://doi.org/10.1016/S0378-1097(02)00647-X .). En algunos casos, se llegó a relacionar la presencia de una AB, como por ejemplo la tiramina en quesos, con amplificaciones específicas de los genes (Fernández, Flórez, Linares, Mayo y Álvarez, 2006Fernández, M., Flórez, A. B., Linares, D. M., Mayo, B. y Álvarez, M. A. (2006). Early PCR detection of tyramine-producing bacteria during cheese production. Journal of Dairy Research, 73 (3), pp. 318-321. https://doi.org/10.1017/S0022029906001750.). Sin embargo, debido al hecho de que además de la presencia de los productores, hay más factores implicados en la acumulación de AB, pronto comenzaron a desarrollarse otras técnicas que además de la detección permiten la cuantificación e incluso la identificación de los productores. Sin embargo, la técnica de la PCR sigue teniendo mucha utilidad en la selección de cepas que puedan ser utilizadas como cultivos iniciadores en la elaboración de alimentos fermentados (Ladero et al., 2015Ladero, V., Martín, M., Redruello, B., Mayo, B., Flórez, A., Fernández, M. y Álvarez, M. A. (2015). Genetic and functional analysis of biogenic amine production capacity among starter and non-starter lactic acid bacteria isolated from artisanal cheeses. European Food Research and Technology, 241 (3), pp. 377-383. https://doi.org/10.1007/s00217-015-2469-z.). Una simple PCR nos dirá si las cepas candidatas poseen genes de producción de AB y por lo tanto deban ser descartadas como cultivo iniciador, evitando el riesgo de acumulación de AB en grandes cantidades. Existen diversas PCR orientadas a la detección de los genes de producción de histamina, tiramina y putrescina (Costantini, Cersosimo, Prete y García-Moruno, 2006Costantini, A., Cersosimo, M., Prete, V. del y García-Moruno, E. (2006). Production of biogenic amines by lactic acid bacteria: Screening by PCR, thin-layer chromatography, and high-performance liquid chromatography of strains isolated from wine and must. Journal of Food Protection, 69 (2), pp. 391-396. https://doi.org/10.4315/0362-028X-69.2.391.; Fernández, Linares y Álvarez, 2004Fernández, M., Linares, D. M. y Álvarez, M. A. (2004). Sequencing of the tyrosine decarboxylase cluster of Lactococcus lactis IPLA 655 and the development of a PCR method for detecting tyrosine decarboxylating lactic acid bacteria. Journal of Food Protection, 67 (11), pp. 2521-2529. https://doi.org/10.4315/0362-028X-67.11.2521.; Ladero, Rattray et al., 2011Ladero, V., Rattray, F. P., Mayo, B., Martín, M. C., Fernández, M. y Álvarez, M. A. (2011). Sequencing and transcriptional analysis of the biosynthesis gene cluster of putrescine-producing Lactococcus lactis. Applied and Environmental Microbiology, 77 (18), pp. 6409-6418. https://doi.org/10.1128/Aem.05507-11.; Marcobal, Rivas, Moreno-Arribas y Muñoz, 2005Marcobal, A., Rivas, B. de las, Moreno-Arribas, M. V. y Muñoz, R. (2005). Multiplex PCR method for the simultaneous detection of histamine-, tyramine-, and putrescine-producing lactic acid bacteria in foods. Journal of Food Protection, 68 (4), pp. 874-878. https://doi.org/10.4315/0362-028X-68.4.874.), que se han aplicado en la selección de cepas iniciadoras en vino, carne o productos lácteos. En productos lácteos hay que destacar el caso de Lactococcus lactis, principal especie utilizada como cultivo iniciador en la elaboración de quesos. Como se ha comentado anteriormente, aunque la capacidad de producción de putrescina se considera una característica de especie, no todas las cepas la presentan debido a su pérdida durante el proceso de domesticación al entorno lácteo. En algunas cepas, la incapacidad de producir putrescina se debe a una secuencia de inserción que interrumpe la transcripción del operón que constituyen los genes responsables de la producción de putrescina, incluido el gen agdi, que es el gen diana de la PCR. Por lo tanto, la presencia del gen agdi en una cepa de Lactococcus lactis es necesaria pero no suficiente para asegurar que produce putrescina (Ladero, Rattray et al., 2011Ladero, V., Rattray, F. P., Mayo, B., Martín, M. C., Fernández, M. y Álvarez, M. A. (2011). Sequencing and transcriptional analysis of the biosynthesis gene cluster of putrescine-producing Lactococcus lactis. Applied and Environmental Microbiology, 77 (18), pp. 6409-6418. https://doi.org/10.1128/Aem.05507-11.). Para evitar este problema se ha diseñado una PCR alternativa a las de detección de agdi utilizada para la selección de iniciadores en otros productos como el vino o la sidra (Coton et al., 2010Coton, M., Romano, A., Spano, G., Ziegler, K., Vetrana, C., Desmarais, C., Lonvaud-Funel, A., Lucas, P. y Coton, E. (2010). Occurrence of biogenic amine-forming lactic acid bacteria in wine and cider. Food Microbiology, 27 (8), pp. 1078-1085. https://doi.org/10.1016/J.Fm.2010.07.012.), en la que los primers, además de detectar la presencia del cluster AGDI permiten comprobar si este está inactivado o no, facilitando el proceso de selección (Ladero, Rattray et al., 2011Ladero, V., Rattray, F. P., Mayo, B., Martín, M. C., Fernández, M. y Álvarez, M. A. (2011). Sequencing and transcriptional analysis of the biosynthesis gene cluster of putrescine-producing Lactococcus lactis. Applied and Environmental Microbiology, 77 (18), pp. 6409-6418. https://doi.org/10.1128/Aem.05507-11.).
El hecho de que existan varias AB con relevancia en la calidad y seguridad alimentaria y de que una misma cepa pueda producir más de una AB implica que hay que realizar varias PCR para confirmar la seguridad de estas cepas. Por ello, diversos autores han diseñado nuevos primers que no solo detectan la presencia de los genes, sino que con el diseño adecuado los tamaños que genera la amplificación de cada uno de los genes se diferencian unos de otros (Marcobal, Rivas, Moreno-Arribas y Muñoz, 2005Marcobal, A., Rivas, B. de las, Moreno-Arribas, M. V. y Muñoz, R. (2005). Multiplex PCR method for the simultaneous detection of histamine-, tyramine-, and putrescine-producing lactic acid bacteria in foods. Journal of Food Protection, 68 (4), pp. 874-878. https://doi.org/10.4315/0362-028X-68.4.874.; Rivas, Marcobal, Carrascosa y Muñoz, 2006Rivas, B. de las, Marcobal, A., Carrascosa, A. V. y Muñoz, R. (2006). PCR detection of foodborne bacteria producing the biogenic amines histamine, tyramine, putrescine, and cadaverine. Journal of Food Protection, 69 (10), pp. 2509-2514.), incluyendo en algún caso un control positivo de amplificación que le da mayor robustez al método (Coton y Coton, 2005Coton, E. y Coton, M. (2005). Multiplex PCR for colony direct detection of Gram-positive histamine- and tyramine-producing bacteria. Journal of Microbiological Methods, 63 (3), pp. 296-304. https://doi.org/10.1016/j.mimet.2005.04.001.; Coton et al., 2010Coton, M., Romano, A., Spano, G., Ziegler, K., Vetrana, C., Desmarais, C., Lonvaud-Funel, A., Lucas, P. y Coton, E. (2010). Occurrence of biogenic amine-forming lactic acid bacteria in wine and cider. Food Microbiology, 27 (8), pp. 1078-1085. https://doi.org/10.1016/J.Fm.2010.07.012.). De esta forma, en una única reacción se puede comprobar la capacidad de una cepa para producir cualquier AB y consecuentemente descartarla para su uso como cultivo iniciador, aumentando la seguridad de los alimentos fermentados.
PCR cuantitativa (qPCR)
Una evolución de estos métodos de detección de la capacidad de producir AB basados en PCR fue su adaptación al uso de la PCR cuantitativa (qPCR). La qPCR añade a las ventajas de la PCR convencional una mayor rapidez -el resultado se puede obtener en menos de dos horas- y la capacidad de cuantificar el número de copias del gen diana (Martínez et al., 2011Martínez, N., Martín, M. C., Herrero, A., Fernández, M., Álvarez, M. A. y Ladero, V. (2011). qPCR as a powerful tool for microbial food spoilage quantification: Significance for food quality. Trends in Food Science & Technology, 22 (7), pp. 367-376. https://doi.org/10.1016/j.tifs.2011.04.004.). Esto nos permite ver la cantidad de productores de AB presentes en un alimento y seguir su evolución a lo largo del proceso de elaboración y conservación hasta su llegada al consumidor, constituyendo una herramienta de prevención, ya que si en las condiciones de conservación o maduración empleadas se observa un aumento de la población de productores de AB se pueden tratar de aplicar las medidas correctoras adecuadas, como reducir la temperatura de almacenamiento, acortar la vida útil, etc… Además, con un adecuado diseño de los primers la qPCR también permite la realización de estudios poblacionales, pudiendo cuantificar la presencia de diferentes especies productoras (Ladero, Calles-Enríquez, Fernández y Álvarez, 2010Ladero, V., Calles-Enríquez, M., Fernández, M. y Álvarez, M. A. (2010). Toxicological effects of dietary biogenic amines. Current Nutrition and Food Science, 6 (2), pp. 145-156. https://doi.org/10.2174/157340110791233256.; Ladero, Cañedo et al., 2012Ladero, V., Cañedo, E., Pérez, M., Cruz Martín, M., Fernández, M. y Álvarez, M. A. (2012). Multiplex qPCR for the detection and quantification of putrescine-producing lactic acid bacteria in dairy products. Food Control, 27 (2), pp. 307-313. https://doi.org/10.1016/j.foodcont.2012.03.024.).
Existen diferentes métodos de qPCR para la detección de bacterias productoras de AB (Bjornsdottir-Butler, Jones, Benner y Burkhardt, 2011aBjornsdottir-Butler, K., Jones, J. L., Benner, R. y Burkhardt, W. (2011a). Development of a real-time PCR assay with an internal amplification control for detection of Gram-negative histamine-producing bacteria in fish. Food Microbiology, 28 (3), pp. 356-363. https://doi.org/10.1016/J.Fm.2010.06.013.; Fernández, Río, Linares, Martín y Álvarez, 2006Fernández, M., Río, B. del, Linares, D. M., Martín, M. C. y Álvarez, M. A. (2006). Real-time polymerase chain reaction for quantitative detection of histamine-producing bacteria: Use in cheese production. Journal of Dairy Science, 89 (10), pp. 3763-3769. https://doi.org/10.3168/jds.S0022-0302(06)72417-1.; Ladero, Calles-Enríquez, Fernández y Álvarez, 2010Ladero, V., Calles-Enríquez, M., Fernández, M. y Álvarez, M. A. (2010). Toxicological effects of dietary biogenic amines. Current Nutrition and Food Science, 6 (2), pp. 145-156. https://doi.org/10.2174/157340110791233256.; Ladero, Cañedo et al., 2012Ladero, V., Cañedo, E., Pérez, M., Cruz Martín, M., Fernández, M. y Álvarez, M. A. (2012). Multiplex qPCR for the detection and quantification of putrescine-producing lactic acid bacteria in dairy products. Food Control, 27 (2), pp. 307-313. https://doi.org/10.1016/j.foodcont.2012.03.024.; Lucas, Claisse y Lonvaud-Funel, 2008Lucas, P. M., Claisse, O. y Lonvaud-Funel, A. (2008). High frequency of histamine-producing bacteria in the enological environment and instability of the histidine decarboxylase production phenotype. Applied and Environmental Microbiology, 74 (3), pp. 811-817. https://doi.org/10.1128/Aem.01496-07.; Podeur et al., 2015Podeur, G., Dalgaard, P., Leroi, F., Prevost, H., Emborg, J., Martinussen, J., Hansen, L. H. y Pilet, M. F. (2015). Development of a real-time PCR method coupled with a selective pre-enrichment step for quantification of Morganella morganii and Morganella psychrotolerans in fish products. International Journal of Food Microbiology, 203, pp. 55-62. https://doi.org/10.1016/j.ijfoodmicro.2015.03.005.) que se han aplicado al estudio de la presencia de productores en pescados, quesos, vinos, sidras o carnes, pudiendo determinarse límites de la presencia de estos productores que supongan un riesgo para la acumulación de concentraciones de AB por encima de los límites recomendados (Bjornsdottir-Butler et al., 2011bBjornsdottir-Butler, K., Jones, J. L., Benner, R. A. y Burkhardt, W. (2011b). Quantification of total and specific gram-negative histamine-producing bacteria species in fish using an MPN real-time PCR method. Food Microbiology, 28 (7), pp. 1284-1292. https://doi.org/10.1016/J.Fm.2011.05.006.; Ladero, Linares, Fernández y Álvarez, 2008Ladero, V., Linares, D. M., Fernández, M. y Álvarez, M. A. (2008). Real time quantitative PCR detection of histamine-producing lactic acid bacteria in cheese: Relation with histamine content. Food Research International, 41 (10), pp. 1015-1019. https://doi.org/10.1016/j.foodres.2008.08.001.; Ladero, Martínez, Martín, Fernández y Álvarez, 2010Ladero, V., Martínez, N., Martín, M. C., Fernández, M. y Álvarez, M. A. (2010). qPCR for quantitative detection of tyramine-producing bacteria in dairy products. Food Research International, 43 (1), pp. 289-295. https://doi.org/10.1016/j.foodres.2009.10.007., Ladero, Coton et al., 2011Ladero, V., Coton, M., Fernández, M., Buron, N., Martín, M. C., Guichard, H., Coton, E. y Álvarez, M. A. (2011). Biogenic amines content in Spanish and French natural ciders: Application of qPCR for quantitative detection of biogenic amine-producers. Food Microbiology, 28 (3), pp. 554-561. https://doi.org/10.1016/J.Fm.2010.11.005.; Nannelli et al., 2008Nannelli, F., Claisse, O., Gindreau, E., Revel, G. de, Lonvaud-Funel, A. y Lucas, P. M. (2008). Determination of lactic acid bacteria producing biogenic amines in wine by quantitative PCR methods. Letters in Applied Microbiology, 47 (6), pp. 594-599. https://doi.org/10.1111/j.1472-765X.2008.02472.x.; Torriani et al., 2008Torriani, S., Gatto, V., Sembeni, S., Tofalo, R., Suzzi, G., Belletti, N., Gardini, F. y Bover-Cid, S. (2008). Rapid detection and quantification of tyrosine decarboxylase gene (tdc) and its expression in gram-positive bacteria associated with fermented foods using PCR-based methods. Journal of Food Protection, 71 (1), pp. 93-101. https://doi.org/10.4315/0362-028X-71.1.93.). Incluso se ha utilizado para determinar la fuente de contaminación de bacterias productoras durante el procesamiento post-maduración de quesos (Ladero et al. 2009Ladero, V., Fernández, M. y Álvarez, M. A. (2009). Effect of post-ripening processing on the histamine and histamine-producing bacteria contents of different cheeses. International Dairy Journal, 19 (12), pp. 759-762. https://doi.org/10.1016/j.idairyj.2009.05.010.).
Electroforesis en geles con gradiente de desnaturalización (DGGE)
La DGGE es una técnica que, gracias a la electroforesis en gradiente de desnaturalización, generalmente originado por un gradiente de urea u otro agente desnaturalizante como pueda ser la temperatura, permite una separación efectiva de fragmentos de ADN de igual tamaño, pero que difieren en su secuencia nucleotídica, permitiendo por tanto diferenciar variantes de un mismo gen. Esta técnica se ha usado ampliamente para ver diferencias y evolución de poblaciones microbianas en distintos ecosistemas, incluyendo alimentos (Cocolin et al., 2007Cocolin, L., Díez, A., Urso, R., Rantsiou, K., Comi, G., Bermaier, I. y Beimfohr, C. (2007). Optimization of conditions for profiling bacterial populations in food by culture-independent methods. International Journal of Food Microbiology, 120 (1-2), pp. 100-109. https://doi.org/10.1016/j.ijfoodmicro.2007.06.015.; Smidt, 2016Smidt, O. de (2016). The use of PCR-DGGE to determine bacterial fingerprints for poultry and red meat abattoir effluent. Letters in Applied Microbiology, 62 (1), pp. 1-8. https://doi.org/10.1111/lam.12505.; Flórez y Mayo, 2006Flórez, A. B. y Mayo, B. (2006). PCR-DGGE as a tool for characterizing dominant microbial populations in the Spanish blue-veined Cabrales cheese. International Dairy Journal, 16 (10), pp. 1205-1210. https://doi.org/10.1016/j.idairyj.2005.11.008.), mediante la separación de fragmentos de PCR correspondientes a genes de valor taxonómico, como puede ser el gen 16S ribosomal. Esta herramienta se puede adaptar a otros genes, como puedan ser los responsables de la producción de AB, lo que permite el estudio de las poblaciones de productores de AB y tener una visión global de las especies implicadas. Aunque no es una técnica cuantitativa, sí permite ver la evolución de los diferentes grupos de productores de AB de forma cualitativa. Tras la electroforesis, se obtienen patrones de bandas que en muchos casos se pueden asignar directamente a especies concretas. Esta técnica se ha empleado en el estudio de la presencia de productores de histamina en queso (Díaz, Ladero, Redruello et al., 2016Díaz, M., Ladero, V., Redruello, B., Sánchez-Llana, E., Río, B. del, Fernández, M., Martín, M. C. y Álvarez, M. A. (2016). A PCR-DGGE method for the identification of histamine-producing bacteria in cheese. Food Control, 63, pp. 216-223. https://doi.org/10.1016/j.foodcont.2015.11.035.), lo que por un lado permitió identificar especies que no se pensaba que pudiesen estar presentes en los quesos, como Tetragenococcus halophilus, y por otro determinar que el principal responsable de la acumulación de histamina en altas concentraciones en queso es Lactobacillus parabuchneri, lo que abrió la puerta al inicio de estudios focalizados en una mejor comprensión de su fisiología y genética para poder diseñar medidas preventivas específicas que impidan el crecimiento y desarrollo de esta especie productora de histamina (Díaz, Ladero, Río et al. 2016Díaz, M., Ladero, V., Río, B. del, Redruello, B., Fernández, M., Martín, M. C. y Álvarez, M. A. (2016). Biofilm-forming capacity in biogenic amine-producing bacteria isolated from dairy products. Frontiers in Microbiology, 7, 591. https://doi.org/10.3389/fmicb.2016.00591.; Díaz et al., 2018Díaz, M., Río, B. del, Sánchez-Llana, E., Ladero, V., Redruello, B., Fernández, M., Martín, M. C. y Álvarez, M. A. (2018). Lactobacillus parabuchneri produces histamine in refrigetared cheese at a temperature-dependent rate. International Journal of Food Science and Technology, 53 (10), pp. 2342-2348. https://doi.org/10.1111/ijfs.13826.; Wuthrich et al., 2017Wuthrich, D., Berthoud, H., Wechsler, D., Eugster, E., Irmler, S. y Bruggmann, R. (2017). The histidine decarboxylase gene cluster of Lactobacillus parabuchneri was gained by horizontal gene transfer and is mobile within the species. Frontiers in Microbiology, 8, 218. https://doi.org/10.3389/fmicb.2017.00218.).
Secuenciación masiva o de nueva generación (NGS)
El desarrollo de las nuevas técnicas ómicas, y más concretamente las técnicas de secuenciación masiva, así como el abaratamiento de las mismas, ha tenido y seguirá teniendo un gran impacto en la microbiología de alimentos y particularmente en calidad y seguridad alimentaria (Mayo et al., 2014Mayo, B., Rachid, C. T., Alegría, A., Leite, A. M., Peixoto, R. S. y Delgado, S. (2014). Impact of next generation sequencing techniques in food microbiology. Current Genomics, 15 (4), pp. 293-309. https://doi.org/10.2174/1389202915666140616233211.; Walsh, Crispie, Claesson y Cotter, 2017Walsh, A. M., Crispie, F., Claesson, M .J. y Cotter, P. D. (2017). Translating omics to food microbiology. Annual Reviews in Food Science and Technology, 8, pp. 113-134. https://doi.org/10.1146/annurev-food-030216-025729.). En el caso concreto del estudio de métodos para la detección de cepas productoras de AB, tiene un gran potencial. Actualmente la secuenciación completa del genoma de las cepas se considera la forma más completa de caracterizar las cepas bacterianas con potencial para ser incluidas en cultivos iniciadores en las fermentaciones alimentarias, ya que por un lado se pueden ver las características tecnológicas relacionadas con el proceso de fermentación y además verificar la seguridad e inocuidad de dichas cepas, incluyendo la capacidad de producir AB. En los últimos años se han secuenciado una gran variedad de cepas productoras de AB de diferentes géneros y especies (Bjornsdottir-Butler, Leon y Benner, 2016Bjornsdottir-Butler, K., Leon, M. S. y Benner, R. A. Jr. (2016). Draft genome sequences of histamine-producing Morganella psychrotolerans strains. Genome Announcements, 4 (5). https://doi.org/10.1128/genomeA.01001-16.; Ladero et al., 2013Ladero, V., Linares, D. M., Río, B. del, Fernández, M., Martín, M. C. y Álvarez, M. A. (2013). Draft genome sequence of the tyramine producer Enterococcus durans strain IPLA 655. Genome Announcements, 1 (3), e00265-13. https://doi.org/10.1128/genomeA.00265-13.; Ladero et al., 2014Ladero, V., Río, B. del, Linares, D. M., Fernández, M., Mayo, B., Martín, M. C. y Álvarez, M. A. (2014). Genome sequence analysis of the biogenic amine-producing strain Lactococcus lactis subsp. cremoris cect 8666 (formerly GE2-14). Genome Announcements, 2 (5), e01088-14. https://doi.org/10.1128/genomeA.01088-14.; Romano, Trip, Lolkema y Lucas, 2013Romano, A., Trip, H., Lolkema, J. S. y Lucas, P. M. (2013). Three-component lysine/ornithine decarboxylation system in Lactobacillus saerimneri 30a. Journal of Bacteriology, 195 (6), pp. 1249-1254. https://doi.org/10.1128/JB.02070-12.; Wuthrich et al., 2017Wuthrich, D., Berthoud, H., Wechsler, D., Eugster, E., Irmler, S. y Bruggmann, R. (2017). The histidine decarboxylase gene cluster of Lactobacillus parabuchneri was gained by horizontal gene transfer and is mobile within the species. Frontiers in Microbiology, 8, 218. https://doi.org/10.3389/fmicb.2017.00218.). Estos estudios se han utilizado para determinar el origen de los genes implicados en la producción de AB (Wuthrich et al., 2017Wuthrich, D., Berthoud, H., Wechsler, D., Eugster, E., Irmler, S. y Bruggmann, R. (2017). The histidine decarboxylase gene cluster of Lactobacillus parabuchneri was gained by horizontal gene transfer and is mobile within the species. Frontiers in Microbiology, 8, 218. https://doi.org/10.3389/fmicb.2017.00218.), para identificar nuevas rutas de producción (Romano et al. 2013Romano, A., Trip, H., Lolkema, J. S. y Lucas, P. M. (2013). Three-component lysine/ornithine decarboxylation system in Lactobacillus saerimneri 30a. Journal of Bacteriology, 195 (6), pp. 1249-1254. https://doi.org/10.1128/JB.02070-12.), o realizar estudios fisiológicos para profundizar en las condiciones que afectan a su acumulación en alimentos (Río et al., 2016Río, B. del, Redruello, B., Martín, M. C., Fernández, M., Jong, A. de, Kuipers, O. P., Ladero, V. y Álvarez, M. A. (2016). Transcriptome profiling of Lactococcus lactis subsp. cremoris CECT 8666 in response to agmatine. Genomics Data, 7, pp. 112-114. https://doi.org/10.1016/j.gdata.2015.12.003.; Pérez et al., 2017Pérez, M., Ladero, V., Río, B. del, Redruello, B., Jong, A. de, Kuipers, O., Kok, J., Martin, M. C., Fernández, M. y Álvarez, M. A. (2017). The relationship among tyrosine decarboxylase and agmatine deiminase pathways in Enterococcus faecalis. Frontiers in Microbiology, 8, p. 2107. https://doi.org/10.3389/fmicb.2017.02107.).
Otra técnica con gran impacto en microbiología de alimentos ha sido la metataxonómica, que mediante la secuenciación masiva de amplicones de regiones variables de genes de interés taxonómico, principalmente el gen de la subunidad 16S ribosomal permite conocer la distribución de especies bacterianas presentes en una muestra (Alegría, Szczesny, Mayo, Bardowski y Kowalczyk, 2012Alegría, A., Szczesny, P., Mayo, B., Bardowski, J. y Kowalczyk, M. (2012). Biodiversity in Oscypek, a traditional Polish cheese, determined by culture-dependent and -independent approaches. Applied and Environmental Microbiology, 78 (6), pp. 1890-1898. https://doi.org/10.1128/AEM.06081-11.). Recientemente, se ha adaptado esta técnica a la amplificación de fragmentos internos de los genes que codifican la tirosina y la histidina descarboxilasas (O’Sullivan et al., 2015O'Sullivan, D. J., Fallico, V., O'Sullivan, O., McSweeney, P. L., Sheehan, J. J., Cotter, P. D. y Giblin, L. (2015). High-throughput DNA sequencing to survey bacterial histidine and tyrosine decarboxylases in raw milk cheeses. BMC Microbiol, 15 (1), 266. https://doi.org/10.1186/s12866-015-0596-0.). Estos estudios han permitido identificar a Enterococcus faecalis como el principal responsable de la producción de tiramina en quesos o corroborar a Lactobacillus parabuchneri como el responsable de la producción de histamina en productos lácteos (O’Sullivan et al., 2015O'Sullivan, D. J., Fallico, V., O'Sullivan, O., McSweeney, P. L., Sheehan, J. J., Cotter, P. D. y Giblin, L. (2015). High-throughput DNA sequencing to survey bacterial histidine and tyrosine decarboxylases in raw milk cheeses. BMC Microbiol, 15 (1), 266. https://doi.org/10.1186/s12866-015-0596-0.), si bien, debido a la necesidad de conocimientos de bioinformática que adapten los métodos y las bases de datos de referencia existentes al caso concreto de la producción de AB, este tipo de estudios son muy escasos.
CONCLUSIONES Top
El hecho de que las AB puedan acumularse en alimentos en altas concentraciones y que la ingesta de estos alimentos contaminados pueda provocar efectos adversos en el consumidor ha hecho que exista un consenso generalizado sobre la necesidad de reducir la presencia de AB en alimentos. La acumulación de estos compuestos tóxicos en alimentos es un problema multifactorial, donde convergen distintas causas, algunas evitables, como la presencia de bacterias Gram negativas en pescados y otras muy difíciles de modificar, ya que son intrínsecas al proceso de elaboración, como es el caso de los alimentos fermentados. Los avances en el conocimiento de la genética de la producción de AB permiten el diseño de nuevas herramientas, más rápidas y específicas, para realizar una correcta detección e identificación de las bacterias responsables de la producción de AB y de esta manera evitar su presencia durante el proceso de fermentación de los alimentos (véase figura 3). En los próximos años, el desarrollo de las técnicas de secuenciación masiva y la adaptación al estudio de las AB de los métodos de análisis disponibles supondrán nuevos avances que contribuirán a la mejora de la calidad y la seguridad de los alimentos que consumimos.
AGRADECIMIENTOSTop
Este trabajo ha sido realizado gracias a la financiación recibida del Ministerio de Economía y Competitividad a través de la Agencia Estatal de Investigación (AGL2016-78708-R; AEI/FEDER, UE), CSIC (CSIC201770E086 and CSIC201870I091) y el Principado de Asturias (IDI/2018/0 00114, cofinanciado con fondos FEDER).
BIBLIOGRAFÍATop
|
| ○ | Alegría, A., Szczesny, P., Mayo, B., Bardowski, J. y Kowalczyk, M. (2012). Biodiversity in Oscypek, a traditional Polish cheese, determined by culture-dependent and -independent approaches. Applied and Environmental Microbiology, 78 (6), pp. 1890-1898. https://doi.org/10.1128/AEM.06081-11 |
| ○ | Álvarez, M. A. y Moreno-Arribas, M. V. (2014). The problem of biogenic amines in fermented foods and the use of potential biogenic amine-degrading microorganisms as a solution. Trends in Food Science & Technology, 39 (2), pp. 146-155. https://doi.org/10.1016/j.tifs.2014.07.007 |
| ○ | Bardocz, S. (1999). Role of biogenic amines-summing up or what is it we do not know?. En: Bardocz, S., Koninkx, J., Grillo, M. y White, A. (eds.). Biogenically active amines in food (vol. III), pp. 1-4. Disponible en https://op.europa.eu/en/publication-detail/-/publication/ed5a2fd4-32fd-48f7-8175-18a3d28e3b5e/language-en/format-PDF/source-search |
| ○ | Bermúdez, R., Lorenzo, J. M., Fonseca, S., Franco, I. y Carballo, J. (2012). Strains of Staphylococcus and Bacillus isolated from traditional sausages as producers of biogenic amines. Frontiers in Microbiology, 3, 151. https://doi.org/10.3389/fmicb.2012.00151 |
| ○ | Bjornsdottir-Butler, K., Jones, J. L., Benner, R. y Burkhardt, W. (2011a). Development of a real-time PCR assay with an internal amplification control for detection of Gram-negative histamine-producing bacteria in fish. Food Microbiology, 28 (3), pp. 356-363. https://doi.org/10.1016/J.Fm.2010.06.013 |
| ○ | Bjornsdottir-Butler, K., Jones, J. L., Benner, R. A. y Burkhardt, W. (2011b). Quantification of total and specific gram-negative histamine-producing bacteria species in fish using an MPN real-time PCR method. Food Microbiology, 28 (7), pp. 1284-1292. https://doi.org/10.1016/J.Fm.2011.05.006 |
| ○ | Bjornsdottir-Butler, K., Leon, M. S. y Benner, R. A. Jr. (2016). Draft genome sequences of histamine-producing Morganella psychrotolerans strains. Genome Announcements, 4 (5). https://doi.org/10.1128/genomeA.01001-16 |
| ○ | Blackwell, B. (1963). Hypertensive crisis due to monoamine-oxidase inhibitors. Lancet, 282 (7313), pp. 849-851. https://doi.org/10.1016/S0140-6736(63)92743-0 |
| ○ | Bodmer, S., Imark, C. y Kneubuhl, M. (1999). Biogenic amines in foods: histamine and food processing. Inflammatory Research, 48 (6), pp. 296-300. https://doi.org/10.1007/s000110050463 |
| ○ | Bover-Cid, S. y Holzapfel, W. H. (1999). Improved screening procedure for biogenic amine production by lactic acid bacteria. International Journal of Food Microbiology, 53 (1), pp. 33-41. https://doi.org/10.1016/S0168-1605(99)00152-X |
| ○ | Bover-Cid, S., Hugas, M., Izquierdo-Pulido, M. y Vidal-Carou, M C. (2001). Amino acid-decarboxylase activity of bacteria isolated from fermented pork sausages. International Journal of Food Microbiology, 66 (3), pp. 185-189. https://doi.org/10.1016/S0168-1605(00)00526-2 |
| ○ | Bover-Cid, S., Schoppen, S., Izquierdo-Pulido, M. y Vidal-Carou, M. C. (1999). Relationship between biogenic amine contents and the size of dry fermented sausages. Meat Science, 51 (4), pp. 305-311. https://doi.org/10.1016/S0309-1740(98)00120-X |
| ○ | Cachaldora, A., Fonseca, S., Franco, I. y Carballo, J. (2013). Technological and safety characteristics of Staphylococcaceae isolated from Spanish traditional dry-cured sausages. Food Microbiology, 33 (1), pp. 61-68. https://doi.org/10.1016/j.fm.2012.08.013 |
| ○ | Calles-Enríquez, M., Eriksen, B. H., Andersen, P. S., Rattray, F. P., Johansen, A. H., Fernández, M., Ladero, V. y Álvarez, M. A. (2010). Sequencing and transcriptional analysis of the Streptococcus thermophilus histamine biosynthesis gene cluster: factors that affect differential hdcA expression. Applied and Environmental Microbiology, 76 (18), pp. 6231-6238. https://doi.org/10.1128/Aem.00827-10 |
| ○ | Cocolin, L., Díez, A., Urso, R., Rantsiou, K., Comi, G., Bermaier, I. y Beimfohr, C. (2007). Optimization of conditions for profiling bacterial populations in food by culture-independent methods. International Journal of Food Microbiology, 120 (1-2), pp. 100-109. https://doi.org/10.1016/j.ijfoodmicro.2007.06.015 |
| ○ | Costantini, A., Cersosimo, M., Prete, V. del y García-Moruno, E. (2006). Production of biogenic amines by lactic acid bacteria: Screening by PCR, thin-layer chromatography, and high-performance liquid chromatography of strains isolated from wine and must. Journal of Food Protection, 69 (2), pp. 391-396. https://doi.org/10.4315/0362-028X-69.2.391 |
| ○ | Coton, E. y Coton, M. (2005). Multiplex PCR for colony direct detection of Gram-positive histamine- and tyramine-producing bacteria. Journal of Microbiological Methods, 63 (3), pp. 296-304. https://doi.org/10.1016/j.mimet.2005.04.001 |
| ○ | Coton, M., Romano, A., Spano, G., Ziegler, K., Vetrana, C., Desmarais, C., Lonvaud-Funel, A., Lucas, P. y Coton, E. (2010). Occurrence of biogenic amine-forming lactic acid bacteria in wine and cider. Food Microbiology, 27 (8), pp. 1078-1085. https://doi.org/10.1016/J.Fm.2010.07.012 |
| ○ | Díaz, M., Ladero, V., Río, B. del, Redruello, B., Fernández, M., Martín, M. C. y Álvarez, M. A. (2016). Biofilm-forming capacity in biogenic amine-producing bacteria isolated from dairy products. Frontiers in Microbiology, 7, 591. https://doi.org/10.3389/fmicb.2016.00591 |
| ○ | Díaz, M., Ladero, V., Redruello, B., Sánchez-Llana, E., Río, B. del, Fernández, M., Martín, M. C. y Álvarez, M. A. (2016). A PCR-DGGE method for the identification of histamine-producing bacteria in cheese. Food Control, 63, pp. 216-223. https://doi.org/10.1016/j.foodcont.2015.11.035 |
| ○ | Díaz, M., Río, B. del, Ladero, V., Redruello, B., Fernández, M., Martín, M. C. y Álvarez, M. A. (2015). Isolation and typification of histamine-producing Lactobacillus vaginalis strains from cheese. International Journal of Food Microbiology, 215, pp. 117-123. https://doi.org/10.1016/j.ijfoodmicro.2015.08.026 |
| ○ | Díaz, M., Río, B. del, Sánchez-Llana, E., Ladero, V., Redruello, B., Fernández, M., Martín, M. C. y Álvarez, M. A. (2016). Histamine-producing Lactobacillus parabuchneri strains isolated from grated cheese can form biofilms on stainless steel. Food Microbiology, 59, pp. 85-91. https://doi.org/10.1016/j.fm.2016.05.012 |
| ○ | Díaz, M., Río, B. del, Sánchez-Llana, E., Ladero, V., Redruello, B., Fernández, M., Martín, M. C. y Álvarez, M. A. (2018). Lactobacillus parabuchneri produces histamine in refrigetared cheese at a temperature-dependent rate. International Journal of Food Science and Technology, 53 (10), pp. 2342-2348. https://doi.org/10.1111/ijfs.13826 |
| ○ | EFSA Panel on BiologicalHazards (BIOHAZ) (2011). Scientific Opinion on risk based control of biogenic amine formation in fermented foods. EFSA Journal, 9 (10), pp. 2393-2486. |
| ○ | Fernández-No, I. C., Bohme, K., Calo-Mata, P. y Barros-Velázquez, J. (2011). Characterisation of histamine-producing bacteria from farmed blackspot seabream (Pagellus bogaraveo) and turbot (Psetta maxima). International Journal of Food Microbiology, 151 (2), pp. 182-189. https://doi.org/10.1016/j.ijfoodmicro.2011.08.024 |
| ○ | Fernández, M., Flórez, A. B., Linares, D. M., Mayo, B. y Álvarez, M. A. (2006). Early PCR detection of tyramine-producing bacteria during cheese production. Journal of Dairy Research, 73 (3), pp. 318-321. https://doi.org/10.1017/S0022029906001750 |
| ○ | Fernández, M., Linares, D. M. y Álvarez, M. A. (2004). Sequencing of the tyrosine decarboxylase cluster of Lactococcus lactis IPLA 655 and the development of a PCR method for detecting tyrosine decarboxylating lactic acid bacteria. Journal of Food Protection, 67 (11), pp. 2521-2529. https://doi.org/10.4315/0362-028X-67.11.2521 |
| ○ | Fernández, M., Linares, D. M., Río, B. del, Ladero, V. y Álvarez, M. A. (2007). HPLC quantification of biogenic amines in cheeses: correlation with PCR-detection of tyramine-producing microorganisms. Journal of Dairy Research, 74 (3), pp. 276-282. https://doi.org/10.1017/S0022029907002488 |
| ○ | Fernández, M., Río, B. del, Linares, D. M., Martín, M. C. y Álvarez, M. A. (2006). Real-time polymerase chain reaction for quantitative detection of histamine-producing bacteria: Use in cheese production. Journal of Dairy Science, 89 (10), pp. 3763-3769. https://doi.org/10.3168/jds.S0022-0302(06)72417-1 |
| ○ | Flórez, A. B. y Mayo, B. (2006). PCR-DGGE as a tool for characterizing dominant microbial populations in the Spanish blue-veined Cabrales cheese. International Dairy Journal, 16 (10), pp. 1205-1210. https://doi.org/10.1016/j.idairyj.2005.11.008 |
| ○ | Garai, G., Dueñas, M. T., Irastorza, A., Maztin-Álvarez, P .J. y Moreno-Arribas, M. V. (2006). Biogenic amines in natural ciders. Journal of Food Protection, 69 (12), pp. 3006-3012. https://doi.org/10.4315/0362-028X-69.12.3006 |
| ○ | Gardini, F., Ozogul, Y., Suzzi, G., Tabanelli, G. y Ozogul, F. (2016). Technological factors affecting biogenic amine content in foods: a review. Frontiers in Microbiology, 7, 1218. https://doi.org/10.3389/fmicb.2016.01218 |
| ○ | Geornaras, I., Dykes, G. A. y von Holy, A. (1995). Biogenic amine formation by poultry-associated spoilage and pathogenic bacteria. Letters in Applied Microbiology, 21 (3), pp. 164-166. https://doi.org/10.1111/j.1472-765X.1995.tb01032.x |
| ○ | Guidi, L. R. y Gloria, M. B. (2012). Bioactive amines in soy sauce: validation of method, occurrence and potential health effects. Food Chemistry, 133 (2), pp. 323-328. https://doi.org/10.1016/j.foodchem.2012.01.033 |
| ○ | Joosten, H. M. y Northolt, M. D. (1989). Detection, growth, and amine-producing capacity of lactobacilli in cheese. Applied and Environmental Microbiology, 55 (9), pp. 2356-2359. |
| ○ | Jorgensen, L. V., Huss, H. H. y Dalgaard, P. (2000). The effect of biogenic amine production by single bacterial cultures and metabiosis on cold-smoked salmon. Journal of Applied Microbiology, 89 (6), pp. 920-934. https://doi.org/10.1046/j.1365-2672.2000.01196.x |
| ○ | Kalac, P. y Krausova, P. (2005). A review of dietary polyamines: Formation, implications for growth and health and occurrence in foods. Food Chemistry, 90 (1-2), pp. 219-230. https://doi.org/10.1016/j.foodchem.2004.03.044 |
| ○ | Kim, S. H., Field, K. G., Morrissey, M. T., Price, R. J., Wei, C. I. y An, H. (2001). Source and identification of histamine-producing bacteria from fresh and temperature-abused albacore. Journal of Food Protection, 64 (7), pp. 1035-1044. https://doi.org/10.4315/0362-028X-64.7.1035 |
| ○ | Ladero, V., Calles-Enríquez, M., Fernández, M. y Álvarez, M. A. (2010). Toxicological effects of dietary biogenic amines. Current Nutrition and Food Science, 6 (2), pp. 145-156. https://doi.org/10.2174/157340110791233256 |
| ○ | Ladero, V., Cañedo, E., Pérez, M., Cruz Martín, M., Fernández, M. y Álvarez, M. A. (2012). Multiplex qPCR for the detection and quantification of putrescine-producing lactic acid bacteria in dairy products. Food Control, 27 (2), pp. 307-313. https://doi.org/10.1016/j.foodcont.2012.03.024 |
| ○ | Ladero, V., Coton, M., Fernández, M., Buron, N., Martín, M. C., Guichard, H., Coton, E. y Álvarez, M. A. (2011). Biogenic amines content in Spanish and French natural ciders: Application of qPCR for quantitative detection of biogenic amine-producers. Food Microbiology, 28 (3), pp. 554-561. https://doi.org/10.1016/J.Fm.2010.11.005 |
| ○ | Ladero, V., Fernández, M. y Álvarez, M. A. (2009). Effect of post-ripening processing on the histamine and histamine-producing bacteria contents of different cheeses. International Dairy Journal, 19 (12), pp. 759-762. https://doi.org/10.1016/j.idairyj.2009.05.010 |
| ○ | Ladero, V., Fernández, M., Calles-Enríquez, M., Sánchez-Llana, E., Cañedo, E., Martín, M. C. y Álvarez, M. A. (2012). Is the production of the biogenic amines tyramine and putrescine a species-level trait in enterococci?. Food Microbiology, 30 (1), pp. 132-138. https://doi.org/10.1016/J.Fm.2011.12.016 |
| ○ | Ladero, V., Fernández, M., Cuesta, I. y Álvarez, M. A. (2010). Quantitative detection and identification of tyramine-producing enterococci and lactobacilli in cheese by multiplex qPCR. Food Microbiology, 27 (7), pp. 933-939. https://doi.org/10.1016/J.Fm.2010.05.026 |
| ○ | Ladero, V., Linares, D. M., Fernández, M. y Álvarez, M. A. (2008). Real time quantitative PCR detection of histamine-producing lactic acid bacteria in cheese: Relation with histamine content. Food Research International, 41 (10), pp. 1015-1019. https://doi.org/10.1016/j.foodres.2008.08.001 |
| ○ | Ladero, V., Linares, D. M., Río, B. del, Fernández, M., Martín, M. C. y Álvarez, M. A. (2013). Draft genome sequence of the tyramine producer Enterococcus durans strain IPLA 655. Genome Announcements, 1 (3), e00265-13. https://doi.org/10.1128/genomeA.00265-13 |
| ○ | Ladero, V., Martín, M., Redruello, B., Mayo, B., Flórez, A., Fernández, M. y Álvarez, M. A. (2015). Genetic and functional analysis of biogenic amine production capacity among starter and non-starter lactic acid bacteria isolated from artisanal cheeses. European Food Research and Technology, 241 (3), pp. 377-383. https://doi.org/10.1007/s00217-015-2469-z |
| ○ | Ladero, V., Martínez, N., Martín, M. C., Fernández, M. y Álvarez, M. A. (2010). qPCR for quantitative detection of tyramine-producing bacteria in dairy products. Food Research International, 43 (1), pp. 289-295. https://doi.org/10.1016/j.foodres.2009.10.007 |
| ○ | Ladero, V., Rattray, F. P., Mayo, B., Martín, M. C., Fernández, M. y Álvarez, M. A. (2011). Sequencing and transcriptional analysis of the biosynthesis gene cluster of putrescine-producing Lactococcus lactis. Applied and Environmental Microbiology, 77 (18), pp. 6409-6418. https://doi.org/10.1128/Aem.05507-11 |
| ○ | Ladero, V., Río, B. del, Linares, D. M., Fernández, M., Mayo, B., Martín, M. C. y Álvarez, M. A. (2014). Genome sequence analysis of the biogenic amine-producing strain Lactococcus lactis subsp. cremoris cect 8666 (formerly GE2-14). Genome Announcements, 2 (5), e01088-14. https://doi.org/10.1128/genomeA.01088-14 |
| ○ | Landete, J. M., Rivas, B. de las, Marcobal, A. y Muñoz, R. (2007). Molecular methods for the detection of biogenic amine-producing bacteria on foods. International Journal of Food Microbiology, 117 (3), pp. 258-269. https://doi.org/10.1016/j.ijfoodmicro.2007.05.001 |
| ○ | Landete, J. M., Rivas, B. de las, Marcobal, A. y Muñoz, R. (2011). PCR methods for the detection of biogenic amine-producing bacteria on wine. Annals of Microbiology, 61 (1), pp. 159-166. https://doi.org/10.1007/s13213-010-0068-6 |
| ○ | Latorre-Moratalla, M. L., Bosch-Fuste, J., Lavizzari, T., Bover-Cid, S., Veciana-Nogués, M. T. y Vidal-Carou, M. C. (2009). Validation of an ultra high pressure liquid chromatographic method for the determination of biologically active amines in food. Journal of Chromatography A, 1216 (45), pp. 7715-7720. https://doi.org/10.1016/j.chroma.2009.08.072 |
| ○ | Latorre-Moratalla, M. L., Bover-Cid, S., Veciana-Nogués, T. y Vidal-Carou, M. C. (2009). Thin-layer chromatography for the identification and semi-quantification of biogenic amines produced by bacteria. Journal of Chromatography A, 1216 (18), pp. 4128-4132. https://doi.org/10.1016/j.chroma.2009.02.045 |
| ○ | Le Jeune, C., Lonvaud-Funel, A., ten Brink, B., Hofstra, H. y van der Vossen, J. M. (1995). Development of a detection system for histidine decarboxylating lactic acid bacteria based on DNA probes, PCR and activity test. Journal of Appllied Bacteriology, 78 (3), pp. 316-326. https://doi.org/10.1111/j.1365-2672.1995.tb05032.x |
| ○ | Linares, D. M., Cruz Martín, M., Ladero, V., Álvarez, M. A. y Fernández, M. (2011). Biogenic amines in dairy products. Critical Reviews in Food Science and Nutrition, 51 (7), pp. 691-703. https://doi.org/10.1080/10408398.2011.582813 |
| ○ | Linares, D. M., Fernández, M., Río, B. del, Ladero, V., Martín, M. C. y Álvarez, M. A. (2012). The tyrosyl-tRNA synthetase like gene located in the tyramine biosynthesis cluster of Enterococcus durans is transcriptionally regulated by tyrosine concentration and extracellular pH. BMC Microbiology, 12 (23). https://doi.org/10.1186/1471-2180-12-23 |
| ○ | Linares, D. M., Río, B. del, Redruello, B., Ladero, V., Martín, M. C., Fernández, M., Ruas-Mediedo, P. y Álvarez, M. A. (2016). Comparative analysis of the in vitro cytotoxicity of the dietary biogenic amines tyramine and histamine. Food Chemistry, 197, pp. 658-663. https://doi.org/10.1016/j.foodchem.2015.11.013 |
| ○ | Lucas, P. M., Claisse, O. y Lonvaud-Funel, A. (2008). High frequency of histamine-producing bacteria in the enological environment and instability of the histidine decarboxylase production phenotype. Applied and Environmental Microbiology, 74 (3), pp. 811-817. https://doi.org/10.1128/Aem.01496-07 |
| ○ | Lucas, P. y Lonvaud-Funel, A. (2002). Purification and partial gene sequence of the tyrosine decarboxylase of Lactobacillus brevis IOEB 9809. FEMS Microbiology Letters, 211 (1), pp. 85-89. https://doi.org/10.1016/S0378-1097(02)00647-X |
| ○ | Maijala, R. L. (1993). Formation of histamine and tyramine by some lactic acid bacteria in MRS-broth and modified decarboxylation agar. Letters in Applied Microbiology, 17 (1), pp. 40-43. https://doi.org/10.1111/j.1472-765X.1993.tb01431.x |
| ○ | Marcobal, A., Rivas, B. de las, Moreno-Arribas, M. V. y Muñoz, R. (2005). Multiplex PCR method for the simultaneous detection of histamine-, tyramine-, and putrescine-producing lactic acid bacteria in foods. Journal of Food Protection, 68 (4), pp. 874-878. https://doi.org/10.4315/0362-028X-68.4.874 |
| ○ | Marcobal, A., Rivas, B. de las, Moreno-Arribas, M. V. y Muñoz, R. (2006). Evidence for horizontal gene transfer as origin of putrescine production in Oenococcus oeni RM83. Applied and Environmental Microbiology, 72 (12), pp. 7954-7958. https://doi.org/10.1128/Aem.01213-06 |
| ○ | Martín, B., Garriga, M., Hugas, M., Bover-Cid, S., Veciana-Nogués, M. T. y Aymerich, T. (2006). Molecular, technological and safety characterization of Gram-positive catalase-positive cocci from slightly fermented sausages. International Journal of Food Microbiology, 107 (2), pp. 148-158. https://doi.org/10.1016/j.ijfoodmicro.2005.08.024 |
| ○ | Martín, M. C., Fernández, M., Linares, D. M. y Álvarez, M. A. (2005). Sequencing, characterization and transcriptional analysis of the histidine decarboxylase operon of Lactobacillus buchneri. Microbiology-Sgm, 151, pp. 1219-1228. https://doi.org/10.1099/mic.0.27459-0 |
| ○ | Martínez, N., Martín, M. C., Herrero, A., Fernández, M., Álvarez, M. A. y Ladero, V. (2011). qPCR as a powerful tool for microbial food spoilage quantification: Significance for food quality. Trends in Food Science & Technology, 22 (7), pp. 367-376. https://doi.org/10.1016/j.tifs.2011.04.004 |
| ○ | Masson, F., Johansson, G. y Montel, M. C. (1999). Tyramine production by a strain of Carnobacterium divergens inoculated in meat-fat mixture. Meat Science, 52 (1), pp. 65-69. https://doi.org/10.1016/S0309-1740(98)00149-1 |
| ○ | Mayo, B., Rachid, C. T., Alegría, A., Leite, A. M., Peixoto, R. S. y Delgado, S. (2014). Impact of next generation sequencing techniques in food microbiology. Current Genomics, 15 (4), pp. 293-309. https://doi.org/10.2174/1389202915666140616233211 |
| ○ | Mayr, C. M. y Schieberle, P. (2012). Development of stable isotope dilution assays for the simultaneous quantitation of biogenic amines and polyamines in foods by LC-MS/MS. Journal of Agricultural and Food Chemistry, 60 (12), pp. 3026-3032. https://doi.org/10.1021/jf204900v |
| ○ | Mercogliano, R., Felice, A. de, Chirollo, C. y Cortesi, M. L. (2010). Production of vasoactive amines during the ripening of Pecorino Carmasciano cheese. Veterinarian Research Communnications, 34, pp. 175-178. https://doi.org/10.1007/s11259-010-9394-1 |
| ○ | Mitar, I., Ljubenkov, I., Rohtek, N., Prkic, A., Andelic, I. y Vuletic, N. (2018). The content of biogenic amines in croatian wines of different geographical origins. Molecules, 23 (10), 2570. https://doi.org/10.3390/molecules23102570 |
| ○ | Nannelli, F., Claisse, O., Gindreau, E., Revel, G. de, Lonvaud-Funel, A. y Lucas, P. M. (2008). Determination of lactic acid bacteria producing biogenic amines in wine by quantitative PCR methods. Letters in Applied Microbiology, 47 (6), pp. 594-599. https://doi.org/10.1111/j.1472-765X.2008.02472.x |
| ○ | Novella-Rodríguez, S., Veciana-Nogués, M. T., Izquierdo-Pulido, M. y Vidal-Carou, M. C. (2003). Distribution of biogenic amines and polyamines in cheese. Journal of Food Science, 68 (3), pp. 750-755. |
| ○ | Novella-Rodríguez, S. N., Veciana-Nogués, M. T., Roig-Sagués, A. X., Trujillo-Mesa, A .J. y Vidal-Carou, M. C. (2004). Evaluation of biogenic amines and microbial counts throughout the ripening of goat cheeses from pasteurized and raw milk. Journal of Dairy Research, 71 (2), pp. 245-252. https://doi.org/10.1017/S0022029904000147 |
| ○ | Novella-Rodríguez, S., Veciana-Nogués, M. T. y Vidal-Carou, M. C. (2000). Biogenic amines and polyamines in milks and cheeses by ion-pair high performance liquid chromatography. Journal of Agricultural and Food Chemistry, 48 (11), pp. 5117-5123. https://doi.org/10.1021/Jf0002084 |
| ○ | O'Sullivan, D. J., Fallico, V., O'Sullivan, O., McSweeney, P. L., Sheehan, J. J., Cotter, P. D. y Giblin, L. (2015). High-throughput DNA sequencing to survey bacterial histidine and tyrosine decarboxylases in raw milk cheeses. BMC Microbiol, 15 (1), 266. https://doi.org/10.1186/s12866-015-0596-0 |
| ○ | Ozdestan, O. y Uren, A. (2010). Biogenic amine content of kefir: A fermented dairy product. European Food Research and Technology, 231 (1), pp. 101-107. https://doi.org/10.1007/s00217-010-1258-y |
| ○ | Pérez, M., Calles-Enríquez, M., Nes, I., Martín, M. C., Fernández, M., Ladero, V. y Álvarez, M. A. (2015). Tyramine biosynthesis is transcriptionally induced at low pH and improves the fitness of Enterococcus faecalis in acidic environments. Applied Microbiology and Biotechnology, 99 (8), pp. 3547-3558. https://doi.org/10.1007/s00253-014-6301-7 |
| ○ | Pérez, M., Ladero, V., Río, B. del, Redruello, B., Jong, A. de, Kuipers, O., Kok, J., Martin, M. C., Fernández, M. y Álvarez, M. A. (2017). The relationship among tyrosine decarboxylase and agmatine deiminase pathways in Enterococcus faecalis. Frontiers in Microbiology, 8, p. 2107. https://doi.org/10.3389/fmicb.2017.02107 |
| ○ | Pinho, O., Pintado, A. I. E., Gomes, A. M. P., Pintado, M. M. E., Malcata, F. X. y Ferreira, I. M. (2004). Interrelationships among microbiological, physicochemical, and biochemical properties of Terrincho cheese, with emphasis on biogenic amines. Journal of Food Protection, 67 (12), pp. 2779-2785. |
| ○ | Podeur, G., Dalgaard, P., Leroi, F., Prevost, H., Emborg, J., Martinussen, J., Hansen, L. H. y Pilet, M. F. (2015). Development of a real-time PCR method coupled with a selective pre-enrichment step for quantification of Morganella morganii and Morganella psychrotolerans in fish products. International Journal of Food Microbiology, 203, pp. 55-62. https://doi.org/10.1016/j.ijfoodmicro.2015.03.005 |
| ○ | Redruello, B., Ladero, V., Cuesta, I., Álvarez-Buylla, J. R., Martín, M. C., Fernández, M. y Álvarez, M. A. (2013). A fast, reliable, ultra high performance liquid chromatography method for the simultaneous determination of amino acids, biogenic amines and ammonium ions in cheese, using diethyl ethoxymethylenemalonate as a derivatising agent. Food Chemistry, 139 (1-4), pp. 1029-1035. https://doi.org/10.1016/j.foodchem.2013.01.071 |
| ○ | Redruello, B., Ladero, V., Río, B. del, Fernández, M., Martín, M. C. y Álvarez, M. A. (2016). Data on recovery of 21 amino acids, 9 biogenic amines and ammonium ions after spiking four different beers with five concentrations of these analytes. Data in Brief, 9, pp. 398-400. https://doi.org/10.1016/j.dib.2016.09.011 |
| ○ | Río, B. del, Binetti, A. G., Martín, M. C., Fernández, M., Magadan, A. H. y Álvarez, M. A. (2007). Multiplex PCR for the detection and identification of dairy bacteriophages in milk. Food Microbiology, 24 (1), pp. 75-81. https://doi.org/10.1016/j.fm.2006.03.001 |
| ○ | Río, B. del, Redruello, B., Linares, D. M., Ladero, V., Fernández, M., Martín, M. C., Ruas-Madiedo, P. y Álvarez, M. A. (2017). The dietary biogenic amines tyramine and histamine show synergistic toxicity towards intestinal cells in culture. Food Chemistry, 218, pp. 249-255. https://doi.org/10.1016/j.foodchem.2016.09.046 |
| ○ | Río, B. del, Redruello, B., Linares, D. M., Ladero, V., Ruas-Madiedo, P., Fernández, M., Martin, M. C. y Álvarez, M. A. (2019). The biogenic amines putrescine and cadaverine show in vitro cytotoxicity at concentrations that can be found in foods. Scientific Reports, 9 (1), e120. https://doi.org/10.1038/s41598-018-36239-w |
| ○ | Río, B. del, Redruello, B., Martín, M. C., Fernández, M., Jong, A. de, Kuipers, O. P., Ladero, V. y Álvarez, M. A. (2016). Transcriptome profiling of Lactococcus lactis subsp. cremoris CECT 8666 in response to agmatine. Genomics Data, 7, pp. 112-114. https://doi.org/10.1016/j.gdata.2015.12.003 |
| ○ | Rivas, B. de las, Marcobal, A., Carrascosa, A. V. y Muñoz, R. (2006). PCR detection of foodborne bacteria producing the biogenic amines histamine, tyramine, putrescine, and cadaverine. Journal of Food Protection, 69 (10), pp. 2509-2514. |
| ○ | Rivas, B. de las, Marcobal, A. y Muñoz, R. (2005). Improved multiplex-PCR method for the simultaneous detection of food bacteria producing biogenic amines. FEMS Microbiology Letters, 244 (2), pp. 367-372. https://doi.org/10.1016/j.femsle.2005.02.012 |
| ○ | Romano, A., Ladero, V., Álvarez, M. A. y Lucas, P. M. (2014). Putrescine production via the ornithine decarboxylation pathway improves the acid stress survival of Lactobacillus brevis and is part of a horizontally transferred acid resistance locus. International Journal of Food Microbiology, 175, pp. 14-19. https://doi.org/10.1016/j.ijfoodmicro.2014.01.009 |
| ○ | Romano, A., Trip, H., Lolkema, J. S. y Lucas, P. M. (2013). Three-component lysine/ornithine decarboxylation system in Lactobacillus saerimneri 30a. Journal of Bacteriology, 195 (6), pp. 1249-1254. https://doi.org/10.1128/JB.02070-12 |
| ○ | Shukla, S., Park, H. K., Kim, J. K. y Kim, M. (2010). Determination of biogenic amines in Korean traditional fermented soybean paste (Doenjang). Food and Chemical Toxicology, 48 (5), pp. 1191-1195. https://doi.org/10.1016/j.fct.2010.01.034 |
| ○ | Silla Santos, M. H. (1996). Biogenic amines: their importance in foods. International Journal of Food Microbiology, 29 (2-3), pp. 213-231. https://doi.org/10.1016/0168-1605(95)00032-1 |
| ○ | Smidt, O. de (2016). The use of PCR-DGGE to determine bacterial fingerprints for poultry and red meat abattoir effluent. Letters in Applied Microbiology, 62 (1), pp. 1-8. https://doi.org/10.1111/lam.12505 |
| ○ | Taylor, S. L. y World Health Organization (1985). Histamine poisoning associated with fish, cheese and other foods. World Health Organization. Report VPH/FOS/85.1. [En línea]. Disponible en https://apps.who.int/iris/handle/10665/66407 |
| ○ | ten Brink, B., Damink, C., Joosten, H. M. y Tveld, J. H. (1990). Occurrence and formation of biologically-active amines in foods. International Journal of Food Microbiology, 11 (1), pp. 73-84. https://doi.org/10.1016/0168-1605(90)90040-C |
| ○ | Torriani, S., Gatto, V., Sembeni, S., Tofalo, R., Suzzi, G., Belletti, N., Gardini, F. y Bover-Cid, S. (2008). Rapid detection and quantification of tyrosine decarboxylase gene (tdc) and its expression in gram-positive bacteria associated with fermented foods using PCR-based methods. Journal of Food Protection, 71 (1), pp. 93-101. https://doi.org/10.4315/0362-028X-71.1.93 |
| ○ | Trip, H., Mulder, N. L., Rattray, F. P. y Lolkema, J. S. (2011). HdcB, a novel enzyme catalysing maturation of pyruvoyl-dependent histidine decarboxylase. Molecular Microbiology, 79 (4), pp. 861-871. https://doi.org/10.1111/j.1365-2958.2010.07492.x |
| ○ | Walsh, A. M., Crispie, F., Claesson, M .J. y Cotter, P. D. (2017). Translating omics to food microbiology. Annual Reviews in Food Science and Technology, 8, pp. 113-134. https://doi.org/10.1146/annurev-food-030216-025729 |
| ○ | Wuthrich, D., Berthoud, H., Wechsler, D., Eugster, E., Irmler, S. y Bruggmann, R. (2017). The histidine decarboxylase gene cluster of Lactobacillus parabuchneri was gained by horizontal gene transfer and is mobile within the species. Frontiers in Microbiology, 8, 218. https://doi.org/10.3389/fmicb.2017.00218 |
| ○ | Yongsawatdigul, J., Choi, Y. J. y Udomporn, S. (2004). Biogenic amines formation in fish sauce prepared from fresh and temperature- abused Indianan chovy (Stolepho rusindicus). Journal of Food Science, 69 (4), pp. 312-319. https://doi.org/10.1111/j.1365-2621.2004.tb06333.x |
|
|
| Recursos en línea |
| ○ | Scombrotoxin (histamine) formation. [En línea]. Disponible en https://www.fda.gov/media/80248/download |
|